海神单胞菌属(Neptunomonas)最初由Hedlund等[1]于1999年定名,代表菌株是从被杂酚油污染的港口沉积物中分离出的食萘海神单胞菌N. naphthovorans NAG-2N-126,其具有降解多环芳烃的能力。海神单胞菌属为革兰氏阴性,杆状,有鞭毛,氧化酶和过氧化氢酶呈阳性[2]。迄今为止,共有8个海神单胞菌属菌种被发现和鉴定,分别为N. naphthovorans[1]、N. concharum[3]、N. japonica[4]、N. acidivorans[5]、N. antarctica[6]、N. qingdaonensis[7]、N. marina[8]和N. phycophila[9],其中6个菌种已经完成全基因组测序[2, 4, 7-10]。
芳香族化合物是分子中至少含有一个离域键的环状化合物,如苯、萘、蒽、菲及其衍生物等。芳香族化合物的结构较为稳定、不易分解,且普遍存在“三致效应”,在环境中的残留物质进入食物链会对人体健康产生一定的危害[11]。海神单胞菌属的部分菌株可以降解多环芳烃,可能在去除海洋和土壤等环境中的芳香族化合物污染物方面发挥一定作用。Li等[12-13]研究了食萘海神单胞菌对菲的降解,发现添加表面活性剂能够在一定程度上提高菲在培养基中的可溶性,由此提高菲的生物可利用性和菌株对菲的降解速度。目前仅有极少数研究者关注到海神单胞菌对多环芳烃的降解,缺少关于其代谢途径、关键降解酶和降解机理等方面的研究。
聚羟基脂肪酸酯(polyhydroxyalkanoate,PHA)是微生物生长代谢不平衡时在细胞内合成的一种储能用高分子生物材料。PHA具有生物相容性、生物可降解性和生物可再生性等优良特性,在包装材料与组织工程领域具有广阔的应用前景。目前PHA的生产成本高于石油来源的不可降解塑料,限制了其大规模商业化生产和应用开发。研究新型的PHA生产菌种,开发低成本的生产工艺日益受到学术界和工业界的关注。在海神单胞菌中,N. antarctica S3-22能够以果糖为碳源合成聚-3-羟基丁酸酯(polyhydroxybutyrate,PHB),但该菌株的最适生长温度为15 ℃左右,发酵成本高,不利于大规模工业化生产[14]。另一种海神单胞菌N. concharum JCM17730的最适生长温度为37 ℃,对乙酸和丁酸具有较好的利用能力,能够以高浓度丁酸为底物进行不灭菌发酵合成PHB,产量超过15 g/L[15]。考虑到乙酸和丁酸等短链挥发性脂肪酸可利用生物质资源或通过合成气发酵获得[16],其作为发酵碳源替代传统的葡萄糖等淀粉基底物来生产PHA具有良好的应用前景。
目前,关于海神单胞菌在芳香族化合物降解和聚羟基脂肪酸酯生产方面的研究还处于起步阶段,尚需进一步深入探索。本文对海神单胞菌属中6个已测序菌种的基因组数据进行了分析,重点研究了芳香族化合物降解和聚羟基脂肪酸酯代谢途径的关键酶基因在这些菌种中的分布情况,为将来海神单胞菌能够利用廉价底物高效生产聚羟基脂肪酸酯以及开发能够应用于环境治理的微生物制剂提供参考。
1 分析方法 1.1 序列来源海神单胞菌属的基因组信息来自NCBI (National Center for Biotechnology Information)(https://www.ncbi.nlm.nih.gov)。在NCBI数据库中搜索目标菌株的全基因组信息并下载,获得相关菌种的基因组数据,使用软件SnapGene(版本4.3.6)[17]对其保存和分析。
1.2 系统发育树分析在NCBI数据库中检索海神单胞菌属相关菌种的16S rRNA序列并下载,利用软件MEGA(版本7.0)对系统发育树进行分析,使用Neighbor-Joining法对16S rRNA序列构建相应的系统发育树[18]。
1.3 基因组平均核苷酸一致性分析利用工具EzGenome(http://www.ezbiocloud.net/ezgenome/ani)分析海神单胞菌相关菌种的全基因组序列信息的平均核苷酸一致性(average nucleotide identity, ANI)[19]。
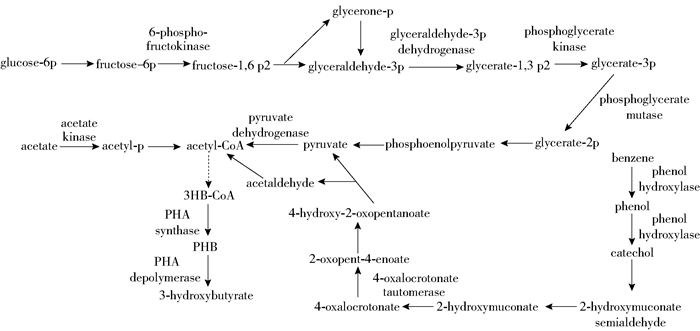
1.4 代谢途径关键基因分析海神单胞菌属中可能的碳源代谢、芳香族化合物代谢及聚羟基脂肪酸酯代谢的路径如图 1所示。利用SnapGene软件和NCBI网站的Blast功能,分析代谢途径中关键酶的编码基因在海神单胞菌属各菌种基因组中的分布情况。
|
图 1 海神单胞菌属中可能的代谢通路 Fig.1 The possible metabolic pathways in the Neptunomonas genus |
碳源代谢是微生物的主要产能途径。本文分析的碳源代谢途径包括糖酵解途径(glycolytic pathway)、Entner-Doudoroff(ED)途径、磷酸戊糖途径、乙酸利用和丙酮酸代谢途径等。相关酶包括6-磷酸果糖激酶(6-phosphofructokinase)、丙酮酸激酶(pyruvate kinase)、3-磷酸甘油醛脱氢酶(glyceraldehyde 3-phosphate dehydrogenase)、磷酸甘油酸激酶(phosphoglycerate kinase)、磷酸甘油酸变位酶(phosphoglycerate mutase)、乙酸激酶(acetate kinase)和丙酮酸脱氢酶(pyruvate dehydrogenase)等。
1.4.2 芳香族化合物降解途径芳香族化合物在有氧条件下的生物降解一般经过邻苯二酚或其衍生物,再开环形成直链分子进入中心代谢途径分解。相关酶主要包括苯酚羟化酶(phenol hydroxylase)、2-辛烷基-6-甲氧基苯酚羟化酶(2-octaprenyl-6-methoxyphenol hydroxylase)、4-羟基苯丙酮酸双加氧酶(4-hydroxyphenylpyruvate dioxy-genase)、邻苯二酚2, 3-双加氧酶(catechol dioxyge-nase)、4-羧基黏糠酸内酯脱羧酶(4-carboxymuconolactone decarboxylase)、羧基黏糠酸内酯脱羧酶(carboxymuconolactone decarboxylase)、4-草酰乙酸脱羧酶(4-oxaloacetate decarboxylase)、4-草酰巴豆酸酯互变异构酶(4-oxalocrotonate tautomerase)等。
1.4.3 聚羟基脂肪酸酯代谢途径聚羟基脂肪酸酯代谢的关键酶是PHA合成酶(PHA synthase)和PHA降解酶(PHA depolymerase),前者催化酯酰辅酶A单体的聚合,后者催化PHA的胞内分解再利用。
2 结果与讨论 2.1 海神单胞菌属的基本特征和基因组测序分析在目前已发现和鉴定的8个海神单胞菌属菌种中,N. qingdaonensis为好氧菌,其余均为兼性厌氧菌;多数菌种的最适生长温度为20~30 ℃,有的菌种具有在细胞内积累聚-3-羟基丁酸酯的能力(表 1)。不同菌种之间生长温度和需氧情况的差异可能与其长期的生存环境和生活习性有关。
| 下载CSV 表 1 海神单胞菌属的表型特征 Table 1 Phenotypic characteristics of the Neptunomonas genus |
海神单胞菌属的6个菌种已经有公开的基因组序列。N. phycophila包括3个菌株,N. marina包含1个菌株,其余4个菌种(N. concharum、N. japonica、N. qingdaonensis和N. antarctica)均包含2个菌株。本文对其中的11个菌株的基因组序列进行分析,结果如表 2所示。可以看出,这些菌株的基因组大小差距较大:N. antarctica DSM22306的基因组最大,为4 569 005 bp;N. concharum JCM17730的基因组最小,为3 561 992 bp。基因组GC含量在43.7%~50.5%(摩尔分数)之间。对已测序的海神单胞菌的全基因组序列进行平均核苷酸一致性分析,结果如表 3所示。海神单胞菌属各个菌种之间的平均核苷酸一致性均在69%以上,同一菌种不同菌株的平均核苷酸一致性均在90%以上。基因组大小的差异暗示了海神单胞菌属不同菌株在基因功能上可能存在差别。对基因组最小的N. concharum和较大的N. qingdaonensis P10-2-4、N. antarctica S3-22进行特有基因分析,结果如表 4所示。发现N. qingdaonensis和N. antarctica在氨基酸、糖类和脂质的转运与代谢、细胞壁和细胞膜合成以及信号传递等方面的特有基因较多。
| 下载CSV 表 2 海神单胞菌属的基因组概况 Table 2 Genome overview of the Neptunomonas genus |
| 下载CSV 表 3 海神单胞菌属基因组平均核苷酸一致性分析 Table 3 Analysis of the average nucleotide identity of the genome of Neptunomonas genus |
| 下载CSV 表 4 海神单胞菌属3个菌种特有基因的同源蛋白簇(COG)功能分类 Table 4 COG function classification of the specific genes of three strains of Neptunomonas genus |
对海神单胞菌属已经鉴定的8个菌种的模式菌株的16S rRNA序列和作为外群的大肠杆菌(Escherichia coli)的16S rRNA序列构建系统发育树,结果如图 2所示。在海神单胞菌属内,N. qingdaonensis、N. japonica和N. antarctica的亲缘关系较近,N. acidivorans和N. phycophila的亲缘关系较近,而N. marina和同一属内其他菌种的亲缘关系较远。由16S rRNA构建系统发育树得到的亲缘关系结论与基因组平均核苷酸一致性的分析结果类似。

|
大肠杆菌ATCC 11775 (X80725)用作外群;括号中给出GenBank登录号;系统发育树中树枝节点处的数字代表重复1 000次计算得到的自展值;标尺0.02表示每个位点的核苷酸取代数。下同。 图 2 海神单胞菌属的16S rRNA系统发育树 Fig.2 A 16S rRNA-based phylogenetic tree of the Neptunomonas genus |
基于基因组测序数据对海神单胞菌属各代表菌株的碳源代谢途径相关基因进行分析,糖酵解途径、Entner-Doudoroff途径、磷酸戊糖途径、乙酸和丙酮酸代谢途径的部分关键酶的基因座位如表 5所示。海神单胞菌属中,已测序的菌种都含有糖酵解途径中的关键酶,包括丙酮酸激酶、3-磷酸甘油醛脱氢酶、磷酸甘油酸激酶和磷酸甘油酸变位酶等(图 1),表明糖酵解途径在海神单胞菌属中普遍存在。但是,部分海神单胞菌的磷酸转移酶系统不完整,例如N. concharum JCM17730和N. japonica JAMM 1380仅有PtsH蛋白,缺少细胞膜上运输葡萄糖的PtsG蛋白,提示它们不具有利用葡萄糖的能力。根据基因组测序数据可知,磷酸戊糖途径的关键酶葡萄糖-6-磷酸脱氢酶、Entner-Doudoroff途径的关键酶磷酸葡萄糖酸脱水酶只在N. antaratica S3-22和N. phycophila Scap09中被发现。另外,基因组分析表明,海神单胞菌普遍缺少木糖转运和利用的关键基因,表明海神单胞菌属的糖代谢普遍不活跃。
| 下载CSV 表 5 海神单胞菌属中碳源代谢相关酶的基因座位 Table 5 The locus tags of enzymes related to carbon metabolism in the Neptunomonas genus |
乙酸代谢途径的关键酶乙酸激酶和丙酮酸代谢途径的丙酮酸脱氢酶在已测序的菌种中也都有发现,提示海神单胞菌属具有利用乙酸的能力。细菌以乙酸为唯一碳源时,一方面通过三羧酸循环将乙酰辅酶A彻底氧化以提供能量,另一方面启动乙醛酸循环合成琥珀酸等中间体用于生物合成反应,基因组测序分析发现了乙醛酸循环相关的异柠檬酸裂解酶和苹果酸合成酶的存在。此外,丙酸代谢相关的丙酰辅酶A合成酶也普遍存在,且有多个拷贝。
2.3.2 芳香族化合物代谢相关基因基于基因组测序数据对海神单胞菌属各代表菌株的芳香族化合物代谢途径相关基因进行分析,其中涉及的部分关键酶的基因座位如表 6所示。海神单胞菌属普遍含有苯、苯酚、苯甲酸的降解途径,与大多数细菌一样采用双加氧酶进行多环芳烃的降解,而真菌往往采用单加氧酶进行降解。苯和苯酚通过苯酚羟化酶催化生成邻苯二酚[20],该酶在两个菌种N. concharum JCM17730和N. japonica JAMM 1380中存在。苯甲酸由苯甲酸双加氧酶催化生成邻苯二酚,但该酶只在菌种N. phycophila Scap09中被发现。
| 下载CSV 表 6 海神单胞菌属中芳香化合物降解相关酶的基因座位 Table 6 The locus tags of enzymes related to degradation of aromatic compounds in the Neptunomonas genus |
邻苯二酚有两条降解途径:邻位降解和间位降解。邻位降解是将邻苯二酚转化为3-酮己二酸,再与辅酶A缩合,分解为琥珀酸和乙酰辅酶A,进入三羧酸循环被彻底氧化,涉及的酶包括邻苯二酚1, 2-双加氧酶、黏糠酸环异构酶、黏糠酸内酯异构酶等,该途径关键酶的编码基因只在N. phycophila Scap09中被发现。间位降解是将邻苯二酚降解为丙酮酸和乙酰辅酶A,进入三羧酸循环氧化分解,其中涉及的酶有邻苯二酚-2, 3-双加氧酶、4-草酰乙酸脱羧酶、4-草酰巴豆酸酯互变异构酶等(图 1)。在大部分海神单胞菌属的菌种中能找到间位降解途径所涉及到的酶,例如N. concharum JCM17730和N. japonica JAMM 1380都有完整的将苯酚降解为丙酮酸和乙酰辅酶A的间位降解代谢途径,但缺少邻位降解途径。目前,已报道的好氧条件下降解多环芳烃的菌属超过100个,研究主要集中在Pseudomonas、Sphingomonas、Burkholderia、Mycobacterium和Rhodococcus等少数几个菌属,降解途径中关键酶如多环芳烃羟基化双加氧酶、邻苯二酚双加氧酶等也有结构解析[21]。但是,对于包括海神单胞菌在内的其他菌属的报道较少,因此还需深入研究不同菌属对芳香族化合物的降解途径、降解机理以及在自然条件下菌群迁移转化和协同作用等。
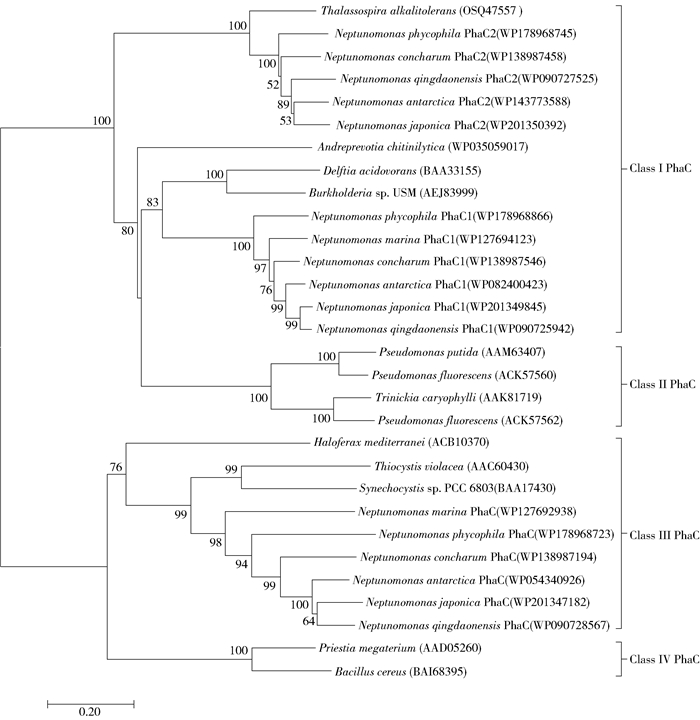
2.3.3 聚羟基脂肪酸酯代谢相关基因碳源可通过多种代谢途径转化为羟基脂酰辅酶A单体,由PHA合成酶催化聚合得到PHA[22-23]。PHA合成酶是PHA生物合成途径中最为关键的酶,其底物特异性决定了脂酰辅酶A单体的类型,也是控制PHA聚合物单体组成和分子量的关键因素[22]。微生物在碳源缺乏时,PHA降解酶催化细胞内积累的PHA分解再利用。本文对海神单胞菌属各代表菌株的PHA合成酶和PHA降解酶的基因组测序数据进行了分析,相关基因座位见表 7。对PHA合成酶序列进行系统发育树分析,结果见图 3。由表 7可以看出,海神单胞菌属的全部菌种均含有2~3个PHA合成酶,并且在基因组上分散排布;海神单胞菌属普遍含有PHA降解酶基因,表明其具有对PHA降解再利用的能力。
| 下载CSV 表 7 海神单胞菌属中聚羟基脂肪酸酯代谢相关酶的基因座位 Table 7 The locus tags of enzymes related to polyhydroxyalkanoate metabolism in the Neptunomonas genus |
|
图 3 海神单胞菌属的PHA合成酶及其他代表性PHA合成酶的发育树 Fig.3 Phylogenetic tree of the PHA synthases from the Neptunomonas genus and other representative strains |
PHA合成酶根据结构、亚基组成和底物特异性,可分为4种类型。其中,Ⅰ型合成酶由一个亚基组成,Ⅱ型合成酶是包含2个亚基的异源二聚体[24]。在一些PHA合成菌株中,PHA合成酶与代谢途径中的其他基因紧密相连。例如,罗氏真养菌H16(Ralstonia eutropha H16)中PHA聚合酶与硫解酶、乙酰乙酰辅酶A还原酶的基因组成一个操纵子,紫色色杆菌(Chromobacterium violaceum)中PHA聚合酶与硫解酶的基因组成一个操纵子。由图 3发现3个PHA合成酶中2个为Ⅰ型、1个为Ⅲ型,Ⅲ型合成酶PhaC和PhaE的基因在基因组中前后相连。海神单胞菌中PHA合成酶的分布情况与海生杆菌属(Marinobacterium)[25]较为相似,两个菌属同为海洋螺菌科,具有较近的亲缘关系。
3 结论本文对海神单胞菌属已鉴定的6个菌种的特征进行了系统总结,并对其基因组测序数据进行了分析。研究发现海神单胞菌属的菌种普遍具有降解芳香族化合物的相关基因,理论上可将苯、苯酚和苯甲酸等通过以邻苯二酚为中间体的间位降解途径进行分解,可在降解芳香族化合物方面发挥作用。另外,海神单胞菌属具有完整的糖酵解和乙酸利用途径;普遍含有聚羟基脂肪酸酯的合成酶和降解酶基因,表明其具有潜在的合成聚羟基脂肪酸酯的能力。相关分析结果可以为海神单胞菌属的进一步代谢工程改造用于生产PHA提供参考。
| [1] |
HEDLUND B P, GEISELBRECHT A D, BAIR T J, et al. Polycyclic aromatic hydrocarbon degradation by a new marine bacterium, Neptunomonas naphthovorans gen. nov., sp. nov.[J]. Applied and Environmental Microbiology, 1999, 65(1): 251-259. DOI:10.1128/AEM.65.1.251-259.1999 |
| [2] |
PU N, LI W, LI Z J. Complete genome sequence of Neptunomonas concharum JCM17730T: an acetate assimilating bacterium isolated from a dead ark clam[J]. Marine Genomics, 2020, 53: 100754. DOI:10.1016/j.margen.2020.100754 |
| [3] |
LEE H W, SHIN N R, LEE J, et al. Neptunomonas concharum sp. nov., isolated from a dead ark clam, and emended description of the genus Neptunomonas[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt_11): 2657-2661. DOI:10.1099/ijs.0.037473-0 |
| [4] |
MIYAZAKI M, NOGI Y, FUJIWARA Y, et al. Neptunomonas japonica sp. nov., an Osedax japonicus symbiont-like bacterium isolated from sediment adjacent to sperm whale carcasses off Kagoshima, Japan[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(4): 866-871. DOI:10.1099/ijs.0.65509-0 |
| [5] |
YANG S H, SEO H S, LEE J H, et al. Neptunomonas acidivorans sp. nov., isolated from sediment, and emended description of the genus Neptunomonas[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt_11): 3650-3654. DOI:10.1099/ijs.0.064253-0 |
| [6] |
ZHANG X Y, ZHANG Y J, YU Y, et al. Neptunomonas antarctica sp. nov., isolated from marine sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(8): 1958-1961. DOI:10.1099/ijs.0.017756-0 |
| [7] |
LIU A, ZHANG X Y, CHEN C X, et al. Neptunomonas qingdaonensis sp. nov., isolated from intertidal sand[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63(Pt_5): 1673-1677. DOI:10.1099/ijs.0.041970-0 |
| [8] |
CHEN W M, HSIEH T Y, SHEU S Y. Neptunomonas marina sp. nov., isolated from seawater[J]. Archives of Microbiology, 2019, 201(8): 1053-1060. DOI:10.1007/s00203-019-01671-7 |
| [9] |
FROMMLET J, GUIMARÃES B, SOUSA L, et al. Neptunomonas phycophila sp. nov. isolated from a culture of Symbiodinium sp., a dinoflagellate symbiont of the sea anemone Aiptasia tagetes[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(Pt_3): 915-919. DOI:10.1099/ijs.0.000039 |
| [10] |
RONG J C, LIU M, LI Y, et al. Insight into the genome sequence of a sediment-adapted marine bacterium Neptunomonas antarctica S3-22T from Antarctica[J]. Marine Genomics, 2016, 25: 29-31. DOI:10.1016/j.margen.2015.11.006 |
| [11] |
厉舒祯, 邓晔, 张照婧, 等. 生物降解芳香族化合物的分子检测技术研究进展[J]. 中国环境科学, 2019, 39(6): 2577-2587. LI S Z, DENG Y, ZHANG Z J, et al. Advances in molecular detection on aromatic bioremediation[J]. China Environmental Science, 2019, 39(6): 2577-2587. (in Chinese) DOI:10.3969/j.issn.1000-6923.2019.06.041 |
| [12] |
LI J L, CHEN B H. Effect of nonionic surfactants on biodegradation of phenanthrene by a marine bacteria of Neptunomonas naphthovorans[J]. Journal of Hazardous Materials, 2009, 162(1): 66-73. DOI:10.1016/j.jhazmat.2008.05.019 |
| [13] |
LI J L, BAI R. Effect of a commercial alcohol ethoxylate surfactant (C11-15E7) on biodegradation of phenanthrene in a saline water medium by Neptunomonas naphthovorans[J]. Biodegradation, 2005, 16(1): 57-65. DOI:10.1007/s10531-004-0429-1 |
| [14] |
LIU X J, ZHANG J, HONG P H, et al. Microbial production and characterization of poly-3-hydroxybutyrate by Neptunomonas antarctica[J]. PeerJ, 2016, 4: e2291. |
| [15] |
PU N, HU P, SHI L L, et al. Microbial production of poly(3-hydroxybutyrate) from volatile fatty acids using the marine bacterium Neptunomonas concharum[J]. Bioresource Technology Reports, 2020, 11: 100439. DOI:10.1016/j.biteb.2020.100439 |
| [16] |
杨昊. 代谢工程大肠杆菌利用乙酸合成丙酮与异丙醇的途径优化[D]. 上海: 华东理工大学, 2019. YANG H. Optimization of the synthesis pathway of acetone and isopropanol from acetate by metabolically engineered Escherichia coli[D]. Shanghai: East China University of Science and Technology, 2019. (in Chinese) |
| [17] |
DAIGNEAULT B, VILARINO M, RAJPUT S, et al. 79 CRISPR gene editing in bovine zygotes-mutation confirmation by integration of protein expression and DNA sequencing analyses[J]. Reproduction, Fertility and Development, 2019, 31(1): 165. |
| [18] |
KUMAR S, STECHER G, TAMURA K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 20106, 33(7): 1870-1874. |
| [19] |
LEE I, KIM Y O, PARK S C, et al. OrthoANI: an improved algorithm and software for calculating average nucleotide identity[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2): 1100-1103. DOI:10.1099/ijsem.0.000760 |
| [20] |
KILPI S, BACKSTRÖM V, KORHOLA M. Degradation of catechol, methylcatechols and chlorocatechols by Pseudomonas sp. HV3[J]. FEMS Microbiology Letters, 1983, 18(1-2): 1-5. DOI:10.1111/j.1574-6968.1983.tb00438.x |
| [21] |
黄勇. 细菌好氧降解多环芳烃上游途径基因多样性研究[D]. 北京: 清华大学, 2015. HUANG Y. Diversity and organization of genes involved in the upper pathway of PAHs degradation[D]. Beijing: Tsinghua University, 2015. (in Chinese) |
| [22] |
PU N, WANG M R, LI Z J. Characterization of polyhydroxyalkanoate synthases from the marine bacterium Neptunomonas concharum JCM17730[J]. Journal of Biotechnology, 2020, 319: 69-73. DOI:10.1016/j.jbiotec.2020.06.002 |
| [23] |
LI Z J, SHI Z Y, JIAN J, et al. Production of poly(3-hydroxybutyrate-co-4-hydroxybutyrate) from unrelated carbon sources by metabolically engineered Escherichia coli[J]. Metabolic Engineering, 2010, 12(4): 352-359. DOI:10.1016/j.ymben.2010.03.003 |
| [24] |
ZOU H B, SHI M X, ZHANG T T, et al. Natural and engineered polyhydroxyalkanoate (PHA) synthase: key enzyme in biopolyester production[J]. Applied Microbiology and Biotechnology, 2017, 101: 7417-7426. DOI:10.1007/s00253-017-8485-0 |
| [25] |
王梦汝, 席威, 李正军. 海生杆菌属的基因组测序数据分析[J]. 生物工程学报, 2020, 36(12): 2695-2706. WANG M R, XI W, LI Z J. Analysis of the genome sequencing data of the Marinobacterium genus[J]. Chinese Journal of Biotechnology, 2020, 36(12): 2695-2706. (in Chinese) |








