癌症是现代医学难以攻克的重疾,其中肺癌的死亡率高居所有癌症死亡率的榜首,分别占男性癌症死亡病例的23%和女性癌症死亡病例的22%[1]。肺癌的低生存率的主要原因在于57%的患者诊断即晚期,其5年生存率只有8%。然而,如果通过计算机断层扫描(CT)进行早期筛查,及时发现肺结节病灶区域,则能够将患者5年存活率提高至57%[1]。因此使用CT图像进行分析和诊断是早期肺癌筛查的重要策略,而肺癌筛查中结节的大小被认为是评估其恶性程度的最重要的线索[2]。准确的结节分割可为后期计算结节收缩体积提供准确的计量结果,同时为药物治疗提供参考信息[3]。
传统的肺结节分割方法多为经典的图像处理方法,通常基于区域生长的方法或形态学操作。Dehmeshki等[4]较早地使用了基于对比度的区域生长方法来分割近球形结节;Kubota等[5]基于局部强度值来确定每个体素属于结节的可能性,再使用区域生长方法将结节从背景中分离出来。然而以上两种区域生长方法均存在初始种子点选择的问题。针对这个问题,Gu等[6]首先提出了将用户点击作为初始种子点的区域生长方法来分割近胸膜结节;而后Song等[7]通过滑降算法搜索各点邻域梯度的最小值,据此获得初始种子点,再由区域生长方法从像素灰度约束和生长距离约束的角度确定肺部病变区域。对于区域生长方法,其最终的分割效果与其种子点及约束策略的选择密切相关,而由于肺结节形态的多样性,很难设定一个对所有类型的结节都适用的策略。基于形态学操作的方法在普适性上面临着相似的困境。Kostis等[8]针对3种不同形态类型的肺结节,包括界限分明的、血管化的和近球形的肺结节,相应地开发了形态学分割方案。Mukhopadhyay[9]将肺结节根据其核心强度分布划分为两类,并使用不同的形态学操作移除局部附着的胸膜表面和血管,达到分割肺结节的目的。尽管这些基于形态学操作的方法快速且易于实现,但是由于结节的大小不同,形态算子的大小难以设置,且在非固体结节的分割上效果欠佳。
以上这些传统的图像处理技术无法提供足够鲁棒和准确的肺结节分割结果。深度学习的发展为医学影像处理领域带来了新思路,如高强等[10]提出了一种基于组归一化与最近邻插值网络(U-net with group normalization and nearest interpolation, GNNI U-net)模型用以分割左心室轮廓,张泽中等[11]提出了一种基于多尺度输入与特征融合的多输入融合网络(multi-input-fusion net, MIFNet)模型用以分割胃癌病理图像。在有关肺结节分割的任务中,Shakibapour等[12]采用的基于特征提取的方法在性能上有了明显提升,他们在已检测到的结节中提取特征数据空间,将一组有关强度和形状的特征向量聚类,最后根据位置和纹理分割不同类型的结节。深度学习的快速崛起,使得深度学习方法在图像分割任务中成为当前较受关注的一种技术[13]。Long等[14]在2015年提出的全卷积网络(fully convolutional networks, FCN)结构实现了对图像进行像素级的分类,从而解决了语义级别的图像分割问题。Ronneberger等[15]提出了U-net网络结构,随后在医学图像处理领域得到广泛使用。Wang等[16-17]提出一种半自动的集中聚焦卷积神经网络和多视图深度卷积神经网络,它们可以更好地分割颈胸结节,但是在性能上仍有改善的空间,特别是在对于小型结节的分割表现上。Liu等[18]先将肺结节区域截取为不同尺度的连续斑块,再输入基于残差块的双路径网络模型中来分割肺结节。Usman等[19]提出一种半自动的自适应提取肺结节斑块的方法,使得肺结节的分割精度有了一定的提升。
为了取得更加精准的分割效果,本文提出一种全局注意力机制引导的肺结节精准分割方法。区别于Attention机制[20],本文所提方法利用区域建议网络(region proposal network, RPN)[21]在全局的基础上提取肺结节的感兴趣区域,并将其转换为注意力权重,与原图一起作为后续分割模型的输入。相较于完全依赖CT图像本身进行的肺结节分割让模型自主学习而带来的不确定性,本文方法为分割模型提供了更为明确的注意范围。
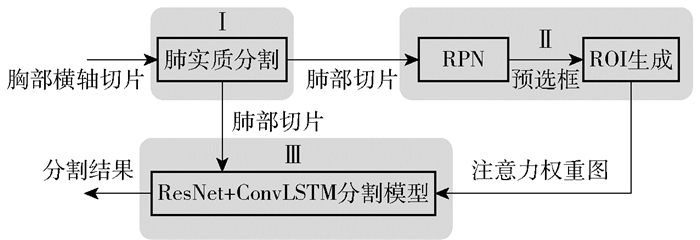
1 肺结节分割方法注意力机制目前在自然语言处理与图像识别领域中运用广泛,传统的做法是在网络训练的同时加入Attention模块,使得模型能够在训练过程中更加聚焦在待识别的物体区域。但由于训练过程中网络参数分布会随着训练的迭代发生改变,可能使得Attention模型注意力偏移。本文提出一种将注意力机制以权重值的形式叠加在原始图像上进行深度分割网络训练的方法,通过将注意力机制与待分割样本进行融合,能够使模型在训练过程中更加聚焦,同时注意力不会随着网络参数分布的变化而发生偏移,使得对肺结节的分割精度得到显著提升。本文提出的肺结节分割算法的框架如图 1所示,可以大致分为如下3个部分:
|
图 1 所提方法的顶层框架 Fig.1 Top-level framework of the proposed method |
1) 采用基于阈值的方法从完整的胸部影像横轴切片中分割出肺实质区域;
2) 利用区域建议网络在肺实质切片上提取肺结节感兴趣区域,进一步缩小分割网络关注的范围,并将其转换为权重注意图;
3) 融合肺实质切片和注意力权重图作为输入,使用基于残差网络(ResNet)与卷积长短期记忆网络(convolutional long short-term memory,ConvLSTM)[22]结构的分割模型实现肺结节分割。
图 1中模块Ⅰ、Ⅱ尽可能地缩小了分割模型所需要关注的范围,减轻了后续分割模型的任务;同时模块Ⅱ基于肺实质切片的全局信息为分割模型提供了注意力权重,显著提高了分割精度。
1.1 基于阈值的肺实质分割所采用的原始数据为整个胸腔的CT影像,而分割肺结节仅需要关注肺部区域,因此在开展肺结节分割工作之前,首先要对数据进行预处理,将肺实质与其他组织分隔开。本文使用基于阈值的分割方法,并辅以形态学修补来达到分割肺实质的目的,其流程及效果如图 2所示。选取靠近图像中心并包含肺实质区域的平均像素值作为阈值,结合K均值聚类方法,将原始胸腔影像轴向切片二值化,使得可以通过此阈值将肺和比肺叶密度更高的组织区分开来。然后对得到的二值分割结果进行形态学修补,经过腐蚀、清理背景、膨胀、选取两个最大的连通区域并填充等一系列操作后,得到完整肺叶轮廓的mask图。最后将原胸腔影像轴向切片与得到的肺部mask图进行叠加计算,分割得到肺实质。

|
图 2 肺实质分割流程及效果示意图 Fig.2 The process and effect of lung parenchyma segmentation |
Ren等[21]创新性地提出了RPN结构,使得Faster R-CNN在目标检测领域的表现引起人们的广泛关注。RPN在Faster R-CNN中用来提供候选检测框的建议,它将特征图上的每个像素在原图上的映射视作锚点,以每个锚点为中心生成若干个固定大小及比例的锚点框,然后对所有的锚点框进行分类及位置回归,再经过筛选即可得到区域的建议结果。
虽然肺结节的大小分布跨度较大,但相对于整个肺实质仍然属于较小目标,尤其是小结节一直是肺结节检测及分割的难题。若直接在肺实质切片的全局上进行分割,则存在大量的背景区域及干扰,很难取得较高的精度。因此本文创新性地将RPN融合在肺结节分割的整体方案中,在肺实质切片的基础上单独训练RPN网络,使其可以根据肺实质切片的全局信息提供肺结节的区域建议。利用越可能包含肺结节区域、RPN会在此区域相应地输出越密集的预选框建议的事实,开发算法将RPN输出转换为全局注意力权重。在保留细节信息的前提下,利用RPN进一步缩小分割网络的关注范围的同时,生成的注意力权重图也使得后续的分割工作更加聚焦,帮助其取得更高的分割精度。
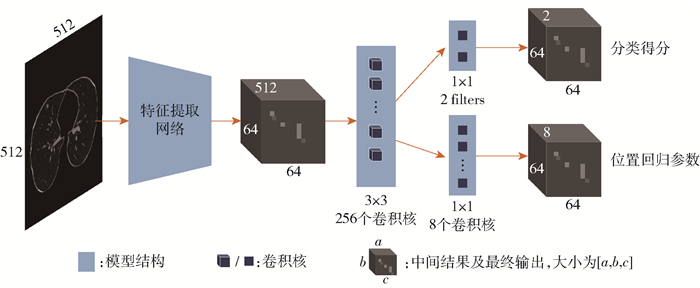
本文所采用的RPN模型如图 3所示。模型设定以每个锚点为中心,生成长边长度序列为[8, 16]、长短边长度比为1 ∶1的锚点框,即每个锚点对应两个锚点框。在模型中输入大小为(512×512)的肺实质切片后,首先经由特征提取网络提取图像中的深层特征;接着在得到的特征空间上滑动3×3的窗口,将该特征图转换为512维的特征向量;最后分别通过两个并列的1×1卷积层,得到RPN的两个输出如下:(1)分类得分,即每个锚点框区域包含肺结节的概率;(2)位置回归参数,对每个锚点框的位置和大小进行一次回归修正。其中特征提取网络采用Res-Net50结构,由于肺结节属于小目标,过深的模型结构会导致特征图太小而丢失有效信息,所以剔除了Res-Net50中最后一个残差模块。
|
图 3 RPN模型结构示意图 Fig.3 RPN model structure diagram |
从原始数据集提取了964张含肺结节的肺实质图片,经过数据扩增(水平翻转与垂直翻转),最后共得到2 892张图片,将其中2 603张图片组成训练集,剩下289张图片组成测试集。训练时,特征提取网络(Res-Net50)先导入在ImageNet数据集上预训练的权重,然后特征提取网络与后续结构作为一个整体进行训练回归。模型训练时使用Adam优化器,学习率设定为1×10-5,最终迭代训练了600轮次。
从RPN模型中得到的输出是对所有锚点框的评价,需要对其进行处理并筛选。首先剔除不合法(范围超出边界)的锚点框,再对其进行非极大值抑制(non-maximum suppression,NMS)[21],得到的预选框将用于构成最终的区域建议(ROI)。非极大值抑制算法将锚点框按分类得分降序排列,每次取集合中得分最高的锚点框纳入最终结果,遍历剩下的锚点框,剔除与选择的锚点框重叠度(IOU值)大于阈值的锚点框,以此标准迭代直到初始集合为空。此时得到的结果仅是可能为肺结节区域的预选框,不能直接用于后续的分割工作。因此本文将这些预选框转换为热图,将热图与原图融合后再作为分割模型的输入。初始时建立一张空白图片,遍历所有预选框,根据式(1)计算每个像素点的注意力权重。
| $ W(i, j)=W(i, j)+\left\{w_{\min } \frac{\sqrt{\left(i-b_{i}\right)^{2}+\left(j-b_{j}\right)^{2}}}{\sqrt{2} r}+\right.\\ \left.w_{\max }\left(1-\frac{\sqrt{\left(i-b_{i}\right)^{2}+\left(j-b_{j}\right)^{2}}}{\sqrt{2} r}\right)\right\} $ | (1) |
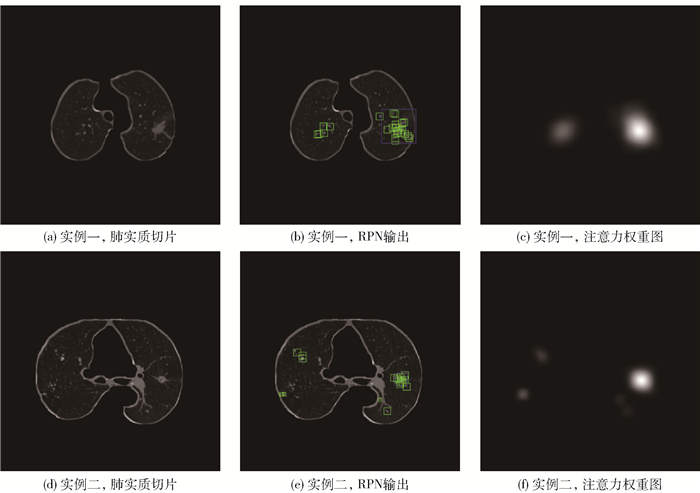
式中,W(i,j)为坐标(i,j)的像素点的注意力权重(灰度值,初始为0);(bi,bj)为预选框范围内某像素点的坐标;r为以(bi,bj)为中心的权重半径;(wmin,wmax)为权重变化范围,为了保证小结节的分割效果,针对较小尺寸的预选框设定更大的权重。即遍历预选框中的每个像素点,以其为中心叠加一个半径为r、灰度值渐变的圆,便可以根据RPN输出的预选框生成注意力权重图。经实验论证,设定r=20,较大预选框的权重变化范围为(0,50)、较小预选框为(0,120),可以在后续分割中取得不错的效果。得到最终的处理效果如图 4所示。可以发现真实存在肺结节的区域有着更密集的预选框,采用此方法将使得越可能存在肺结节的区域其注意力权重图对应位置的权重越高。这样不仅能根据得到的RPN输出缩小后续分割模型的关注区域,更能为这些区域赋予不同的权重,帮助后续的模型获得更好的分割效果。
|
蓝色框为真值框,绿色框为筛选后的模型输出。 图 4 RPN模型输出展示 Fig.4 RPN model output display |
本文所采用的分割模型沿用U-Net经典的编码-解码结构。其中编码基于ResNet结构,而解码使用ConvLSTM的内存机制来融合从编码器的不同阶段提取的特征,如图 5所示。由于得到的注意力权重图可以帮助分割网络将注意区域大幅缩小,所以此处将编码结构尽量精简,防止丢失更多的浅层细节信息。

|
图 5 分割模型结构示意图 Fig.5 Schematic diagram of the segmentation model structure |
在编码阶段,从ResNet的顶部一直到底部,在每个残差模块上都推导出一个特征,这些特征可以看作由粗到细的尺度序列。最深的特征具有低分辨率、高感受野的特点,更加考虑全局的图像信息;而上层的特征有着高分辨率和有限的感受野,包含更多的细节信息。
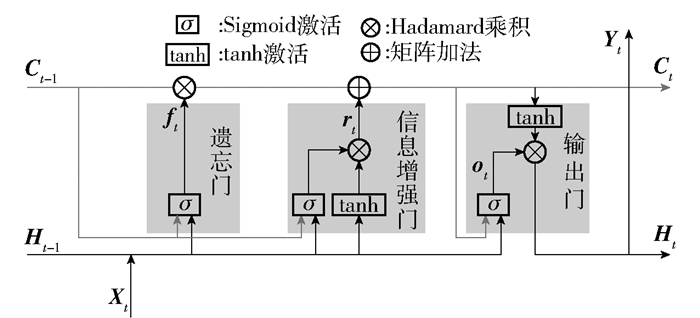
解码阶段使用的ConvLSTM发挥粗-细融合机制的作用。ConvLSTM将原LSTM[23]的权值计算方式替换为卷积操作,使得其不仅能够得到时序关系,还能够像卷积层一样提取空间特征。如图 6所示,Ct-1与Ht-1为上一个单元的输出,Xt为当前状态下的输入,Ct与Ht则为当前单元的输出;每个Conv-LSTM单元中有3个门控机制,遗忘门对上一个节点传进来的输入进行选择性忘记,信息增强门对当前状态的输入有选择性地进行增强,输出门决定哪些会被当成当前状态的输出。ConvLSTM的状态更新公式如式(2)~(6)所示。
| $ \boldsymbol{f}_{t}=\sigma\left(\boldsymbol{w}_{f} *\left[\boldsymbol{H}_{t-1}, \boldsymbol{X}_{t}\right]+\boldsymbol{b}_{f}\right) $ | (2) |
| $ \begin{array}{l} \boldsymbol{r}_{t}=\sigma\left(\boldsymbol{w}_{r 1} *\left[\boldsymbol{H}_{t-1}, \boldsymbol{X}_{t}\right]+\boldsymbol{b}_{r 1}\right) \times \tanh \left(\boldsymbol{w}_{r 2} *\right. \\ \left.\left[\boldsymbol{H}_{t-1}, \boldsymbol{X}_{t}\right]+\boldsymbol{b}_{r 2}\right) \end{array} $ | (3) |
| $ \boldsymbol{o}_{t}=\sigma\left(\boldsymbol{w}_{0} *\left[\boldsymbol{H}_{t-1}, \boldsymbol{X}_{t}\right]+\boldsymbol{b}_{0}\right) $ | (4) |
| $ \boldsymbol{C}_{t}=\boldsymbol{f}_{t} \otimes \boldsymbol{C}_{t-1}+\boldsymbol{r}_{t} $ | (5) |
| $ \boldsymbol{H}_{t}=\boldsymbol{o}_{t} \otimes \tanh \boldsymbol{C}_{t} $ | (6) |
|
图 6 一个ConvLSTM单元的结构示意图 Fig.6 Schematic diagram of the structure of a ConvLSTM unit |
式中,σ表示卷积操作,*表示点乘操作,⊗表示Hadamard乘积。本文在解码阶段采用ConvLSTM对上层传递的特征与编码阶段残差块输出的特征进行融合,这样的机制使得解码模块首先接受最深层的特征图,从而更加关注全局信息,随着解码过程的推进,不断加入浅层次的细节信息,使其起到一个根据上下文分割的功能[24]。当在处理肺结节这种具有较浅的高分辨率特征的目标时,这种结构预期可以取得更优异的效果。
本文使用来自肺图像数据库联盟(LIDC)和图像数据库资源倡议(IDRI)的公共数据集。LIDC-IDRI数据集采用了4位放射科医师的标注,而每个医师的标注之间都存在着差异。针对此问题,本文采用50%一致性准则来生成训练真值,即当一个像素同时被至少两个医师标注时,才将其纳入此结节的真值标注范围。同时,由于数据集中的肺结节尺寸相差较大(分布见表 1),为了保证网络的泛化能力,将数据按结节半径划分子集,各自打乱,每个子集都按照相同比例再次划分,再分别加入训练集、验证集和测试集中,这样就保证了训练时各个子集的数据分布相同。最终训练集、验证集和测试集所包含的数据量分别为1 949、491和269。模型训练使用Adam优化器,学习率设定为1×10-4,以二进制交叉熵函数(binary_cross entropy)作为损失函数,最终迭代训练60轮次。
| 下载CSV 表 1 肺结节大小的数据分布 Table 1 Data distribution of lung nodule size |
LIDC-IDRI数据集中每个实例扫描的薄片厚度在0.6~5.0 mm之间,且都由4位经验丰富的胸部放射科医师进行诊断标注,并将详细的结节信息如边界坐标、恶性程度等记录到XML文件中。数据集包含3种类型的病灶,即非结节、小结节(r < 3 mm)和大结节(r≥3 mm)。在本文研究中只考虑大结节。
以dice相似度系数D(dice similarity coefficient, DSC)作为主要的评价标准来评估本文方法的分割效果。此外,为了保证评价的稳健性,还采用正预测值V(PPV)和灵敏度Se(SEN)作为辅助评价参数。DSC、PPV、SEN的定义如式(7)~(9)所示。
| $ \begin{aligned} &D=\frac{2 \times S(G \cap P)}{S(G)+S(P)} \end{aligned} $ | (7) |
| $ \begin{aligned} &S_{\mathrm{e}}=\frac{S(G \cap P)}{S(G)} \end{aligned} $ | (8) |
| $ \begin{aligned} &V=\frac{S(G \cap P)}{S(P)} \end{aligned} $ | (9) |
式中,G表示真值,P表示模型的分割结果,S表示以像素为单位计算得到的面积。
2 结果与讨论首先在肺结节的10个特征上评估本文方法的分割效果,对比论证所提方法在应对形态多样的肺结节时是否具有普适性。然后以DSC为标准,将本文方法的分割精度与其他5种方法以及4位人类专家的标注结果进行了比较。
2.1 多种形态的肺结节分割结果由于肺结节大小不一,尺寸分布跨度较大,为了评估本文方法是否对各个尺寸的肺结节都普遍适用,将测试集按结节大小划分子集,独立评估每个子集的分割效果,如表 2所示。可以看出,本文所提方法对不同大小的肺结节都能取得不错的分割效果,其中对较大结节的分割效果更为优秀。
| 下载CSV 表 2 在不同大小的结节上的分割表现 Table 2 Segmentation performance on nodules of different sizes |
在LIDC-IDRI数据集中对标注的每个肺结节都用了9个特征来描述,例如calcification(钙化)、sphericity(球形度)、spiculation(毛刺)等,还针对每个特征标注了范围在1~6的得分,用于区分同一特征的不同程度或情况。根据每个特征的评分将测试集划分为不同子集,独立评估本文方法在各个子集中的表现。表 3中展示了各项评估参数,可以看出,采用本文方法处理所有类型的结节都能得到相似且精度较高的性能,证明了本文方法在各种肺结节形态上的普适性,也从侧面反映了方法的鲁棒性。
| 下载CSV 表 3 在不同特征的肺结节上的分割表现 Table 3 Segmentation performance on lung nodules with different characteristics |
首先对本文方法进行了消融实验。在采用相同的训练数据集划分、相同的训练参数的前提下,分别训练了3种不同组成的模型,使它们各自在肺实质分割图的基础上进行肺结节分割。这3种模型组成分别为直接使用U-Net结构、直接使用Res-Net+ConvLSTM结构、先通过RPN获得注意力权重再经由U-Net结构分割(RPN+ U-Net),将所得到的分割效果与本文方法进行对比,结果如表 4所示。可以看出在同等条件下,无论在Res-Net+ConvLSTM模型或U-Net模型之前是否先获取注意力权重来引导分割,Res-Net+ConvLSTM结构取得的平均Dice值均高于U-Net结构,表明Res-Net+ConvLSTM在肺结节分割上的表现略优于U-Net。此外,对比表 4还能发现,通过在分割网络之前添加注意力权重模块从而引导分割模型进行分割,能使分割精度得到大幅提升。其中U-Net模型搭配注意力权重模块,分割精度相比于仅使用U-Net模型提升了15.29%;本文方法的分割精度相比仅使用Res-Net+ConvLSTM模型提升了13.22%,充分证明了先通过RPN获得注意力权重图、再据此引导Res-Net+ConvLSTM模型进行分割的方法的有效性。
| 下载CSV 表 4 消融实验对比 Table 4 Comparison of ablation experiments |
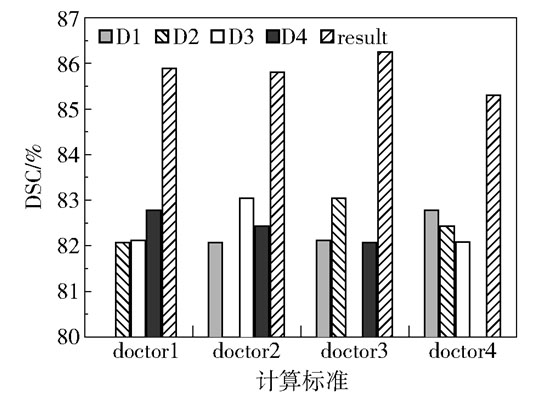
将本文方法的分割结果与4位医生的标注进行一致性比较,结果如图 7所示。可以看出,不论以哪位医生为标准,本文方法的分割结果都比其他几位医生的标注更接近标准结果。最后通过计算得出本文方法的分割结果与每位医生标注之间的DSC平均值±标准差为(85.81±0.39)%,而4位医生之间的相应值为(82.42±0.41)%。本文方法不仅DSC均值更高,且标准差也更低,这说明所提方法的分割结果比人类专家的标注更接近真值,且更具有稳定性。
|
doctorN表示以第N位医生的标注为标准,比较其他几位医生(分别以D1、D2、D3、D4表示)以及本文方法的分割结果(以result表示)与此标准的重合度(测试集上的DSC平均值),同一位医生的比较省略为空。 图 7 本文方法与4位医生之间的一致性比较 Fig.7 Comparison of consistency between this method and four doctors |
最后将本文方法与文献中的肺结节分割方法进行了比较,结果如表 5所示,所有方法都基于LIDC-IDRI数据集进行评估。从表 5可以看出,本文所提方法的DSC平均值能达到89.97%,明显优于现有的分割方法;且本文方法DSC的标准差相较更低,这也能从一定程度上反映出本文方法的稳定性。
| 下载CSV 表 5 各种肺结节分割方法的对比 Table 5 Comparison of various lung nodule segmentation methods |
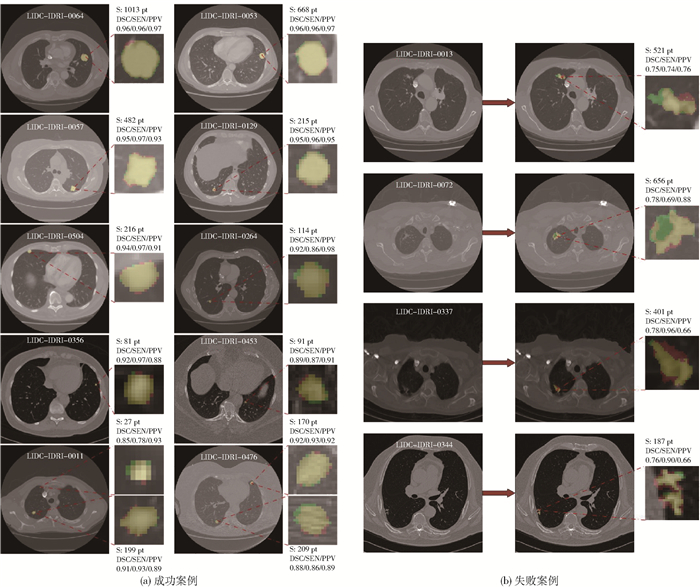
将本文方法整体流程集成之后,处理一张胸腔影像切片平均需要3.04 s,即从输入整张切片到得到肺结节的分割结果所需时间平均为3.04 s,其中全局注意力生成部分耗时较长。本文选取了不同大小的肺结节,从视觉上展示了本文方法的分割效果。图 8由胸腔影像切片及肺结节切片组成,胸腔影像切片上标注了其所属的数据编号,所展示的每个肺结节均标注了其大小及分割结果的参数。从图 8(a)可以看出,本文方法的分割结果已与真值高度重叠。此外,在测试的269份数据中,共有14份分割的Dice值低于0.8,选取了部分具有代表性的案例在图 8(b)中展示。针对这些失败案例进行分析,认为本文方法对于形状非常不规则、边界模糊的肺结节的分割性能有待提高,且针对肺结节与肺实质背景对比度很低的情况,存在分割遗漏的问题。
|
绿色标记肺结节真值区域,红色标记本文方法分割结果,二者重合区域用黄色标记;S为用肺结节区域内像素数量表示的面积,用以表征结节大小。 图 8 肺结节分割效果的视觉展示 Fig.8 Visual display of the segmentation effect of lung nodules |
本文提出一种新的肺结节分割方法,该方法首先对胸部影像横轴切片进行预处理,提取出肺实质区域;再利用RPN网络根据肺实质切片的全局信息生成注意力权重图,在缩小肺结节分割所需关注区域的同时为后续工作提供指导;最后在沿用U-Net经典的编码-解码结构的基础上,结合ResNet与ConvLSTM在注意力权重图的引导下分割出肺结节,实现了从一张完整的胸腔影像横轴切片中自动且精确地分割出肺结节的目的。最终得到的分割精度为89.97%(DSC值),优于现有文献的方法与医生人工标注的效果。对不同大小及类型的肺结节进行的实验评估表明,采用本文方法分割各种形态结节的精度最低为85.52%,最高能达到94.85%,均表现良好,证明了本文方法的普适性与鲁棒性,以及能够达到辅助医师在对肺部疾病的诊断上作出更精确的治疗计划的目的。
| [1] |
SIEGEL R L, MILLER K D, JEMAL A. Cancer statistics, 2020[J]. CA: A Cancer Journal for Clinicians, 2020, 70(1): 7-30. DOI:10.3322/caac.21590 |
| [2] |
KO J P, NAIDICH D P. Computer-aided diagnosis and the evaluation of lung disease[J]. Journal of Thoracic Imaging, 2004, 19(3): 136-155. DOI:10.1097/01.rti.0000135973.65163.69 |
| [3] |
MULSHINE J L, BAER T M. Quantitative imaging tools for lung cancer drug assessment[M]. Hoboken: Wiley, 2008.
|
| [4] |
DEHMESHKI J, AMIN H, VALDIVIESO M, et al. Segmentation of pulmonary nodules in thoracic CT scans: a region growing approach[J]. IEEE Transactions on Medical Imaging, 2008, 27(4): 467-480. DOI:10.1109/TMI.2007.907555 |
| [5] |
KUBOTA T, JEREBKO A K, DEWAN M, et al. Segmentation of pulmonary nodules of various densities with morphological approaches and convexity models[J]. Medical Image Analysis, 2011, 15: 133-154. DOI:10.1016/j.media.2010.08.005 |
| [6] |
GU Y H, KUMAR V, HALL L O, et al. Automated delineation of lung tumors from CT images using a single click ensemble segmentation approach[J]. Pattern Recognition, 2013, 46(3): 692-702. DOI:10.1016/j.patcog.2012.10.005 |
| [7] |
SONG J D, YANG C Y, FAN L, et al. Lung lesion extraction using a toboggan based growing automatic segmentation approach[J]. IEEE Transactions on Medical Imaging, 2016, 35(1): 337-353. DOI:10.1109/TMI.2015.2474119 |
| [8] |
KOSTIS W J, REEVES A P, YANKELEVITZ D F, et al. Three-dimensional segmentation and growth-rate estimation of small pulmonary nodules in helical CT images[J]. IEEE Transactions on Medical Imaging, 2003, 22(10): 1259-1274. DOI:10.1109/TMI.2003.817785 |
| [9] |
MUKHOPADHYAY S. A segmentation framework of pulmonary nodules in lung CT images[J]. Journal of Digital Imaging, 2016, 29(1): 86-103. DOI:10.1007/s10278-015-9801-9 |
| [10] |
高强, 高敬阳, 赵地. GNNI U-net: 基于组归一化与最近邻插值的MRI左心室轮廓精准分割网络[J]. 计算机科学, 2020, 47(8): 213-220. GAO Q, GAO J Y, ZHAO D. GNNI U-net: precise segmentation neural network of left ventricular contours for MRI images based on group normalization and nearest interpolation[J]. Computer Science, 2020, 47(8): 213-220. (in Chinese) |
| [11] |
张泽中, 高敬阳, 赵地. MIFNet: 基于多尺度输入与特征融合的胃癌病理图像分割方法[J]. 计算机应用, 2019, 39(S2): 107-113. ZHANG Z Z, GAO J Y, ZHAO D. MIFNet: pathological image segmentation method for stomach cancer based on multi-scale input and feature fusion[J]. Journal of Computer Applications, 2019, 39(S2): 107-113. (in Chinese) |
| [12] |
SHAKIBAPOUR E, CUNHA A, ARESTA G, et al. An unsupervised metaheuristic search approach for segmentation and volume measurement of pulmonary nodules in lung CT scans[J]. Expert Systems with Applications, 2019, 119: 415-428. DOI:10.1016/j.eswa.2018.11.010 |
| [13] |
GHOSH S, DAS N, DAS I, et al. Understanding deep learning techniques for image segmentation[J]. ACM Computing Surveys, 2019, 52(4): 73. |
| [14] |
LONG J, SHELHAMER E, DARRELL T. Fully convolutional networks for semantic segmentation[C]//IEEE Conference on Computer Vision and Pattern Recognition. Piscataway: IEEE Computer Society, 2015: 3431-3440.
|
| [15] |
RONNEBERGER O, FISCHER P, BROX T. U-Net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer Assisted Intervention. Heidelberg: Springer, 2015: 234-241.
|
| [16] |
WANG S, ZHOU M, LIU Z Y, et al. Central focused convolutional neural networks: developing a data-driven model for lung nodule segmentation[J]. Medical Image Analysis, 2017, 40: 172-183. DOI:10.1016/j.media.2017.06.014 |
| [17] |
WANG S, ZHOU M, GEVAERT O, et al. A multi-view deep convolutional neural networks for lung nodule segmentation[C]//201739 th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC). Jeju: IEEE, 2017: 1752-1755.
|
| [18] |
LIU H, CAO H C, SONG E M, et al. A cascaded dual-pathway residual network for lung nodule segmentation in CT images[J]. Physica Medica, 2019, 63(7): 112-121. |
| [19] |
USMAN M, LEE B D, BYON S S, et al. Volumetric lung nodule segmentation using adaptive ROI with multi-view residual learning[J]. Scientific Reports, 2020, 10: 12839. DOI:10.1038/s41598-020-69817-y |
| [20] |
MNIH V, HEESS N, GRAVES A, et al. Recurrent models of visual attention[C]//Proceedings of the 27th International Conference on Neural Information Processing Systems—Volume 2. Cambridge, USA: MIT Press, 2014: 2204-2212.
|
| [21] |
REN S Q, HE K M, GIRSHICK R, et al. Faster R-CNN: towards real-time object detection with region proposal networks[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(6): 1137-1149. DOI:10.1109/TPAMI.2016.2577031 |
| [22] |
SHI X J, CHEN Z R, WANG H, et al. Convolutional LSTM network: a machine learning approach for precipitation nowcasting[J]. Advances in Neural Information Processing Systems, 2015, 2015: 802-810. |
| [23] |
SUTSKEVER I, VINYALS O, LE Q V. Sequence to sequence learning with neural networks[C]//Proceedings of the 27th International Conference on Neural Information Processing Systems—Volume 2. Cambridge, USA: MIT Press, 2014: 3104-3112.
|
| [24] |
MILLETARI F, RIEKE N, BAUST M, et al. CFCM: segmentation via coarse to fine context memory[C]//International Conference on Medical Image Computing and Computer-assisted Intervention (MICCAI) 2018. Cham: Springer, 2018: 667-674.
|








