2. 秦皇岛环渤海生物产业研究院, 秦皇岛 066000
2. Qinhuangdao Bohai Biological Research Institute, Beijing University of Chemical Technology, Qinhuangdao 066000, China
蜡酯(wax ester)是由长链脂肪酸与长链醇通过酯化反应结合形成的,两部分分子通常都是长链(C16和C18)或超长链(C20或更长)的碳结构。蜡酯在动物、植物和微生物中广泛存在,具有多种重要的生物学功能,对生物的生命活动具有非常重要的意义。植物中的蜡酯主要存在于覆盖初生芽表面的角质层中或者以高浓度的形式积累在一些种子油中,可防止水分流失[1]、紫外辐射[2-3]及致病菌的袭击[3]等;某些动物体内也含有较多的蜡酯,如抹香鲸的鲸油可传递声音、调节浮力等[3-4];各种细菌通过积累蜡酯将其作为主要的碳源和能量储存物质[3, 5]。由于蜡酯具有多种多样的功能和性质,其可作为人类生活中的一种重要的经济油脂,广泛应用于医药、化妆品、食品工业,尤其是能源、化工等领域[6-10]。
近年来,作为商用蜡酯主要来源的动植物蜡酯受有限资源的限制已不能满足不断增长的市场需求。例如商用蜡酯最早的主要来源是鲸蜡油,自从20世纪60年代全球禁止捕鲸以来,蜡酯主要通过昂贵的化学前体合成或者从植物霍霍巴(Simmondsia chinensis)油脂中提取获得,这导致其供应不足且成本高昂,因此仍需寻找廉价的、可持续生产的有效方案。
通过构建人造微生物细胞工厂进行高效生物制造,已被科学界及工业界认可。生物体内的蜡酯一般由长链脂肪酸与长链醇在蜡酯合成酶(wax ester synthase, WS)的催化下生成。近年来,利用不同来源的WS高效合成各种蜡酯化合物已经成为研究热点。为了满足生活中对酯类化合物的大量需求,人们尝试从各种生物中寻找WS,期望以此为基础构建一种高效而成本低廉的微生物细胞工厂用于酯类化合物的合成。WS的底物特异性很低[7],人们可以通过调控底物类型及生长条件、菌株改造、使用酶工程等手段使其生产不同类型的产物[8]。例如,生物柴油是一种由短链醇与长链脂肪酸构成的酯类化合物,其成分主要是脂肪酸甲酯和脂肪酸乙酯,目前主要由动植物油脂经化学转酯反应生产,该方法的缺点是生产成本高、易造成环境污染等。为了解决这些问题,近年来,利用WS通过微生物发酵的方法生产生物柴油得到广泛关注,并取得一系列研究进展[7-10],通过催化脂肪酸和相应短链醇的酯化反应,成功创建了生产脂肪酸乙酯、脂肪酸丁酯[7, 10]等不同生物柴油分子的细胞工厂。但是WS的最适底物不是短链醇,这就造成细胞工厂中产物的转化效率非常低,促使人们通过合成生物学或者蛋白质工程等手段来改造WS。
WS是催化生成蜡酯及相关酯类化合物的关键酶,其应用和改造已成为相应菌株构建的技术难题和关键因素。目前,人们已经从多种生物中鉴定和表征了具有蜡酯合成活性的WS,并将其分为两大类。第一类属于典型的WS,它们仅对蜡酯合成具有活性,从哺乳动物(例如人和小鼠)以及高等植物(例如霍霍巴和矮牵牛花)中鉴定并纯化[4, 11],有望提高霍霍巴油等蜡酯的产量;第二类是具有WS和二酰基甘油酰基转移酶(diacylgycerol acyltransferase, DGAT)活性的双功能酶,即WS/DGAT,其首次从贝氏不动杆菌(Acinetobacter baylyi)中鉴定出[12-13],主要在微生物中表达并用于催化脂肪酸和相应短链醇酯化而生成生物柴油分子。通过比较不同WS对不同底物的活性差异及对其活性位点进行改造,可以改变产物的分子组成,使其具有不同的性质和用途。
本文系统论述了微生物、动物和植物体内的蜡酯合成途径及WS的研究进展,包括:不同来源WS的开发及表征,通过酶工程方法对WS的改造和优化以及近年来WS在生物制造领域中的应用。最后探讨了利用WS合成酯类化合物面临的挑战,并对其研究前景进行了展望。
1 蜡酯的合成途径通过对自然界中蜡酯生物合成途径的详细研究,人们发现蜡酯在不同物种中的合成途径略有不同,但其中最后一步都是由WS催化的。
1.1 微生物中蜡酯的合成途径在微生物中,蜡酯是潜在的碳源和储能的化合物。1997年Reiser等[13]在A. baylyi ADP1中发现、鉴定了微生物的蜡酯合成途径。如图 1中A途径所示,其合成途径主要分为3步:第一步,酰基辅酶A(酰基CoA)被酰基CoA还原酶(acyl-CoA reductase, ACR)还原成相应的脂肪醛;第二步,脂肪醛被脂肪醛还原酶(fatty aldehyde reductase)进一步还原成相应的脂肪醇;最后,酰基CoA脂肪醇转移酶(fatty alcohol acyl-CoA transferase)将脂肪醇与酰基CoA缩合,合成相应的蜡酯。其中酰基CoA还原酶、脂肪醛还原酶是依赖于还原型辅酶Ⅱ(NADPH)发生作用的,酰基CoA脂肪醇转移酶随后被命名为蜡酯合成酶。2003年Kalscheuer等[12]又对A. baylyi ADP1中蜡酯合成的关键酶进行了鉴定,证明其同时具有DGAT和WS的活性。Stöveken等[14]于2005年进一步证明了A. baylyi ADP1的蜡酯合成酶除具有DGAT和WS的活性外,还具有单酰甘油酰基转移酶(monoacylglycerol acyltransferase, MGAT)的活性,能够接受多种物质作为替代受体分子,其中对中链醇表现出最高的特异性。
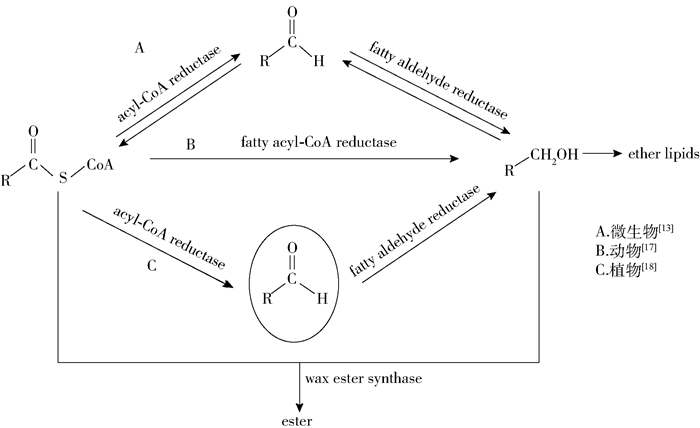
|
图 1 自然界中不同物种蜡酯的生物合成途径 Fig.1 Biosynthetic pathways of wax esters of different species in nature |
Holtzapple等[15]于2007年提出了海杆菌(Marinobacter hydrocarbonoclasticus)DSM 8798中异戊二烯类蜡酯的合成途径。在此途径中,植醇首先经过脱氢酶被氧化为植醛类化合物,然后植醛通过醛脱氢酶被进一步氧化为植酸;同时,植酸被类异戊二烯/酰基CoA合成酶激活为植烷酰基CoA,植烷酰基CoA和植醇通过WS合成类异戊二烯类蜡酯。2017年Tomiyama等[16]鉴定了小眼虫(Euglena gracilis)中的WS/DGAT,证实其参与了厌氧条件下小眼虫体内蜡酯的形成。其体内蜡酯主要是肉豆蔻醇肉豆蔻酸酯(C28)和大量的C26/C27酯类,由中链脂肪酸和C10至C18的醇组成,其中具有C14:0脂肪酸的蜡酯占总蜡酯质量的44%。
1.2 动物中蜡酯的合成途径Cheng等[4, 17]首次表达鉴定了小鼠的两个脂肪酰基CoA还原酶(fatty acyl-CoA reductase, FAR),这两个同工酶FAR1和FAR2分别被其cDNA编码,同时还表达鉴定了小鼠包皮腺中的WS,揭示了哺乳动物中蜡酯的合成途径。如图 1中B途径所示,其蜡酯合成包括由脂肪酰基CoA还原酶和WS催化的两步生物合成途径。该研究证实了脂肪酰基CoA是FAR1的底物,该酶需要NADPH作为辅因子;指明WS在细胞中的表达可以使饱和、不饱和、多不饱和脂肪醇和脂肪酰基CoA合成蜡酯;还发现蜡酯合成酶mRNA在富含皮脂腺的组织(包括眼睑和皮脂腺)中含量最高,而在其他组织中含量较低。
1.3 植物中蜡酯的合成途径植物中脂肪酸的代谢途径主要有两条:第一条是酰基还原途径,可产生蜡酯,蜡酯在生成过程中虽然需要醛类中间体,但并不释放出来(如图 1中C途径所示);另一条是脱羰基化途径,可形成醛、烷烃、仲醇和酮[11]。
霍霍巴油是目前商用蜡酯的主要生物来源,可用于化妆品领域,由油料作物霍霍巴合成。霍霍巴WS的cDNA于2000年在转基因拟南芥(Arabidopsis thaliana)中首次被异源表达,Lardizabal等[19]利用酰基CoA和脂肪醇鉴定了该酶的特性,证明了WS的底物和产物具有亲脂性,还发现WS非常疏水,具有多个潜在的跨膜结构域。Sturtevant等[18]在2020年用霍霍巴基因组中的相应基因详细绘制了酰基-脂质代谢途径,发现已知与蜡酯合成有关的基因(ScFAE1、ScFAR和ScWS)都高度表达,并且明显偏向子叶组织,而三酰甘油合成中的一种末端酶霍霍巴酰基CoA:DGAT1(ScDGAT1)在胚轴组织中显著表达。他们分析标注这些基因,对于霍霍巴植物的基因和代谢的改造具有重要意义。
拟南芥中含有12种WS的同源物,2008年Li等[11]在大肠埃希菌(Escherichia coli)、酿酒酵母(Saccharomyces cerevisiae)中表达并鉴定了拟南芥中蜡酯合成的关键酶WSD1,确定了WSD1参与拟南芥茎蜡酯的形成,并且是催化蜡酯生物合成途径的最后一步的主要酶。该研究发现该酶位于内质网,为今后酶工程的改造以及前体物的有效供应指明了方向。
2 蜡酯合成酶的特性及改造随着越来越多的WS被鉴定,了解这些酶的底物范围和特异性并进行酶工程改造,对工业微生物定向生产蜡酯有着深远影响。本文总结了几种具有代表性的WS的生化特性及活性改造进展。
2.1 蜡酯合成酶的生化特性近年来,各种生物中WS的性质和底物范围被逐步测定,人们发现大多数WS有着广泛的底物范围,且与中长链酯酰CoA和中长链醇有较好的亲和力,这些表征信息对工业化定向生产不同链长的蜡酯有重要意义。
2.1.1 微生物中WS的生物性质海杆菌是革兰氏阴性菌,在M. hydrocarbonoclasticus DSM 8798中,Holtzapple等[15]鉴定了4个编码WS的潜在基因,但是,只有2个基因MhWS1和MhWS2编码的蛋白经E. coli表达纯化后可以在体外检测到WS的活性。随后,Shi等[20]在S. cerevisiae细胞中表达MhWS2,并且在体外检测到MhWS2具有广泛的底物偏好性,发现MhWS2更倾向于催化棕榈酰CoA(C16:0-CoA)和中长链脂肪醇(C10-OH~C16-OH)的反应。Röttig等[21]于2015年在E. coli中表达了MhWS2,但是由于MhWS2无法高效可溶表达,很难得到足够高的纯化量。通过酶动力学分析,他们发现MhWS2对C16:0-CoA显示出很高的亲和性,而短链的酰基CoA则很难被MhWS2转化。Miklaszewska等[22]在2018年详细测定了MhWS2蛋白的底物特异性,他们利用酵母表达,通过干燥的微粒体来测定酶的活性,结果显示该酶在体内和体外均显示出高的WS活性,但是并没有发现其具有DGAT的活性。通过体外实验,检测到MhWS2在较宽的温度范围(45~55 ℃)和pH范围(5.5~10)内表现出较强的活性,且能催化的底物范围较为广泛。在使用C12:0-CoA、C14:1-CoA、C16:0-CoA、C18:0-CoA和C18:1-CoA作为酰基供体时,除了饱和的C20~C24醇利用率很低外,大多数脂肪醇都可以被MhWS2有效地利用。由于在工业生产中生产蜡酯需要调整酵母培养条件,而MhWS2在较宽范围的温度、pH下的高活性表明了其在工业生产中的潜力和优势。
40多年前人们在革兰氏阴性不动杆菌属中首次发现了细菌蜡酯。Kalscheuer等[12]鉴定了不动杆菌中的双功能蜡酯合成酶AtfA(WS/DGAT),揭示了其具有广泛的底物特异性,能够接受C2~C20的饱和、不饱和酰基CoA和C2~C30的线性醇以及支化、环状或芳香醇作为底物。Röttig等[9, 21]通过体外实验测定了纯化的蜡酯合成酶AtfA、AtfA(G355I)和AtfA1(Alcanivorax borkumensis)对不同底物的活性,发现AtfA对底物C16:0-CoA和不饱和醇(C16:1或C18:1)的比活性最高,对较短链的醇(如乙醇或丁醇)的活性较低;当底物浓度超过0.04 mmol/L(C16:0-CoA)或0.2 mmol/L(正十六醇)时,出现了很强的底物抑制作用;而采用正十二醇(浓度在0.6 mmol/L以下)和乙醇则没有发现明显的底物抑制现象,但当使用300 mmol/L以上的甲醇时,该酶的活性急剧下降。对乙醇等短链醇底物和棕榈酰CoA以及较短的酰基CoA底物反应的比较发现,与AtfA相比,AtfA(G355I)对以上底物的活性较低,突变并没有增加其对短链醇底物的比活性,但提高了对乙醇的催化效率和亲和力,这说明AtfA(G355I)对乙醇和短链酰基CoA底物的催化效率和亲和力有着积极的影响。与其他同源酰基转移酶相比,AtfA1的最适底物为C16:0-CoA和C12醇,但AtfA1的总比活性非常低。综上,AtfA仍然具有较大的底物特异性潜质,是生产脂肪酸短链酯的最优选择之一。
迄今为止,关于真核微生物WS的报道一直很少,直到2017年Zhang等[23]首次报道了从海洋生物破囊壶菌(Thraustochytrium roseum)中鉴定的两种双功能WS/DGAT蛋白TrWSD4和TrWSD5,并通过E. coli表达,经体外实验分析了这两种WS/DGAT的偏好性。用C16-OH与酰基受体反应,经动力学特征(米氏常数Km和催化效率kcat/Km,其中kcat为酶的催化常数)比较,发现C12:0-CoA是TrWSD4的最适底物,而C10:0-CoA是TrWSD5的最适底物。在S. cerevisiae中分别表达TrWSD4和TrWSD5后,表达TrWSD4的工程酵母仅可合成蜡酯混合物,而表达TrWSD5的工程酵母还可额外合成棕榈酸乙酯(生物柴油的主要成分),揭示了TrWSD5具有生产生物柴油的潜力。
2.1.2 动植物WS的生物性质Cheng等[4, 17]鉴定了小鼠的WS,对其亚细胞定位进行了表征,通过实时聚合酶链反应(real-time PCR)对mRNA分析并确定了其组织分布。他们从小鼠包皮腺中分离出脂肪酸和脂肪醇,分析了WS对底物的偏好性,发现合成的蜡酯中相应脂肪酸成分的组成丰度大小顺序为:C16:1>C18:1>C16:0> C20:0>C14:0,脂肪醇成分的主要组成为C16:0。Miklaszewska等[24]于2013年在S. cerevisiae中异源表达了小鼠WS,使用转基因酵母的微粒体进行体外酶活测定,发现除了具有WS的活性外,小鼠WS还表现出很低的酰基CoA:DGAT的活性,没有发现具有酰基CoA:MGAT和酰基CoA:甾醇酰基转移酶的活性。他们通过评估小鼠WS对11种酰基CoA与17种脂肪醇底物的组合偏好,发现该酶与中链醇反应时对C14:0-CoA、C12:0-CoA和C16:0-CoA表现出最高的活性(每分钟每毫克微粒体蛋白可分别产生高达5.2、3.4 nmol和3.3 nmol的蜡酯)。在饱和醇与不饱和醇中,该酶对碳链超过18个碳的不饱和醇表现出高活性。在所有测试的醇与C20:0-CoA、C22:1-CoA或Ric-CoA的组合中,小鼠的WS无法充分利用这些底物,且对共轭酰基CoA也没有表现出活性。通过上述酶的体外表征,表明小鼠的WS最适于合成中链蜡酯。
Biester等[25]于2012年在酵母中进行了禽类的几种WS蛋白的表征,比较了不同禽类(包括鸡、鹅、谷仓猫头鹰等)WS的底物特异性,发现所有禽类WS对饱和的中链醇(C10:0-OH~C12:0-OH)和饱和的长链酰基CoA(C14:0-CoA~C18:0-CoA)的活性最高,其中鸡的WS(GgWS1和GgWS2)还对支链酰基CoA保持较高活性;用受测物种的腺体膜作为酶源对不同酰基受体的酶活反应比较证明,所有受测物种的腺体膜均能够催化直链醇(尤其是C12:0-OH)或支链醇(如香叶基香叶醇等)。除WS活性以外,鸡的腺体膜还显示出较高的DGAT活性以及二酯合酶(diester synthase)活性。这些信息对于合成支链蜡酯具有特殊的指导价值。
King等[26]于2007年鉴定了矮牵牛WS,发现其在花瓣中产生低分子量的角质蜡,用14C标记的C16:0-CoA和一系列直链、支化、芳香和萜类化合物醇进行酶活测试,观察到矮牵牛WS对C8-OH~C12-OH醇具有较高活性。Li等[11]于2008年在酵母H1246中表达了拟南芥蜡酯合成酶WSD1,发现当在培养基中添加棕榈酸和链长为C18、C24或C28的醇时表达WSD1的工程菌株皆可合成相应的蜡酯,但是该酶并不催化短链醇(内源醇)生成相应的酯类,表明WSD1可接受长链醇合成长链蜡酯。不同WS的底物偏好性的总结见表 1。
| 下载CSV 表 1 不同WS的底物偏好性 Table 1 Substrate preferences of different wax ester synthases |
在获取WS的晶体及结构之前,研究人员往往通过序列对比、结构预测、随机突变等手段来推断WS的序列和功能的关系,并进行相应改造。2009年Stöveken等[27]对WS/DGAT家族的几种细菌酰基转移酶进行序列对比,发现了高度保守的HHxxxDG基序。他们用亮氨酸取代了不动杆菌的蜡酯合成酶AtfA中该基序的部分序列:第一个组氨酸残基(H132)的取代显著降低了酶活,而第二个组氨酸残基(H133)的取代几乎使酶丧失了活性,当两个组氨酸残基同时被取代时,酶活则完全丧失,说明这两个残基都是AtfA酶发挥活性所必需的。为了进一步了解酶的序列与功能之间的关系,Röttig等[9, 21]对A. baylyi的atfA基因进行了随机诱变、筛选,检测到AtfA的C端Glu15Lys、Trp67Gly、Ala126Asp、Ser374Pro或Gly378Ser/Asp这几种突变严重降低了A. baylyi中的脂质积累和AtfA活性,并据此推测AtfA的C端部分是酶活不可缺少的部分,并猜测C端可能形成二聚化结构域。他们又对AtfA进行了突变得到突变体AtfA(G355I)[21],发现AtfA(G355I)提高了对乙醇等短链醇底物的亲和力。类似地,Barney等[28-29]利用Multalin法通过序列对比推测了影响WS底物偏好性的残基,对应突变了海杆菌(Marinobacter aquaeolei VT8)的蜡酯合成酶(WS1),对突变体进行了活性研究,发现在Leu356、Ala360和Met405上用较大或较小的疏水残基取代,可以使相应突变体对短、支化和芳香醇具有不同的选择性,从而用于合成不同类型的蜡酯。
高精度的蛋白结构有助于阐明WS在微生物中高效催化与专一性的分子机制,进而以高精度预测其催化结构域及活性位点,设计活性、专一性、稳定性改变的突变体。但直到2018年Petronikolou等[30]才得到了WS家族第一个晶体结构,为来自M. aquaeolei VT8的蜡酯合成酶(Ma-WS/DGAT,同源蛋白WS1)。晶体结构显示该酶由两个结构域组成:①N端结构域,由混合的β-折叠(链β1、β4、β5、β6、β11)和一个小的反平行β-折叠(链β2和β3)构成,其中β-折叠(链β1、β4、β5、β6、β11)两侧有4个α螺旋(螺旋α1~α4);②C端结构域,由混合的β-折叠(链β7~β10和β12~β13)组成,一侧覆盖有4个α螺旋(α6、α7、α8、α10),另一侧与螺旋α9和N端结构域相对。通过结构分析发现之前确定的WS家族HHxxxDG保守基序位于N端结构域核心长腔(口袋1)并延伸到C端(口袋2)中的一个回路上;根据两个保守的His的空间位置,发现His136是蛋白发挥催化作用的基团;通过结构数据指出酰基CoA的酰基链结合在N端结构域核心形成的口袋(口袋1)中,并构建了相应WS突变体(口袋1中Gly25→Val和Ala144→Phe),进行底物分析后发现两种突变体最适底物的变化与预测一致,更倾向于链长较短的酰基底物。WS家族晶体结构出现后,人们可以根据结构有目的地进行突变并分析WS的序列和功能之间的联系,WS蛋白的改造将具有较高的可预测性和可控性。
目前人们对WS序列和结构的研究尚处于起步阶段,相关的有效突变位点较少(如表 2所示)。目前仍然是通过简单的生物信息学分析其序列和保守区,或者通过随机突变来改变WS的功能。WS家族第一个晶体结构的出现成为目前WS研究上的一项重大进展[30],然而由于不同WS的蛋白同源性低,通过相似性模拟其他WS的构效关系具有较差的预测精度。未来会出现更多的WS结构,将更好地指导对WS的特异性改造,为微生物细胞工厂高效合成特定成分的蜡酯打下基础。
| 下载CSV 表 2 WS的改造 Table 2 Modifications of wax ester synthases |
目前利用WS的不同底物特异性,同时采取合成生物学手段改变宿主脂类代谢,已开发了多种细胞工厂以使WS应用于不同酯类化合物的生产。
3.1 应用于蜡酯生产利用微生物高效生产具有不同用途的蜡酯一直是人们关注的焦点,为了提高蜡酯的产量,将寻求合成生物学手段实现相应前体物的供应。2018年,Lehtinen等[31]通过在A. baylyi ADP1中异源表达来自Nevskia ramosa的酰基CoA还原酶基因,在菌体中实现过量生产蜡酯合成的异源性前体物脂肪醛,使蜡酯产量达到了0.45 g/L。但是,由于脂肪酸前体供应不足,不动杆菌属能够产生的蜡酯含量很难超过细胞自身干重的15%[32],因此,在其他菌种中表达WS,有可能进一步提升蜡酯产量。
霍霍巴蜡酯是一种性质独特的蜡酯,只能在霍霍巴植物的种子中得到,具有独特的医疗作用,如治疗皮肤感染、加速伤口愈合等[33]。霍霍巴蜡酯是由长链脂肪酰CoA和长链脂肪醇通过WS催化形成的超长链蜡酯,其组成成分主要是碳链为C42:2的蜡酯[34]。在S. cerevisiae中最常见的脂肪酸碳链长度是C16和C18,而超长链脂肪酸的含量很低[35],即使引入WS和FAR后,受自身前体供应的限制,工程酵母仅可合成碳链长度是C28~C36的蜡酯[36],而这些蜡酯在天然霍霍巴蜡酯中占比非常小。Wenning等[37]尝试在S. cerevisiae中异源表达来自M. aquaeolei VT8的FAR和来自S. chinensis的WS基因,同时过表达S. cerevisiae内源β-酮酰基CoA合酶Elo2p,首次在未经外源添加脂肪酸或脂肪醇作为底物的条件下合成了超长链霍霍巴蜡酯(C40:0- WE和C42:0-WE)。在此基础上,他们于2019年将来自Crambe abyssinica的脂肪酸延伸酶(FAE/KCS)和S. cerevisiae内源的脂肪酸脱饱和酶(FAD)Ole1p共表达,使霍霍巴蜡酯的产量相比出发菌提高了2.3倍[38]。目前,通过合成生物学改变宿主中蜡酯前体物碳链的成分和构成,进而通过WS催化合成所需蜡酯的研究还较少,但具备良好的应用及科学价值。
3.2 应用于三酰甘油生产与蜡酯一样,三酰甘油也是一种中性酯,可作为功能性油脂应用于食品、保健领域。由于蜡酯合成酶具有双功能,即WS及DGAT活性,理论上也可用于生成三酰甘油。2019年,Santín等[39]在E. coli中异源表达了来自Thermomonospora curvata的WS/DGAT,使E. coli有了生产三酰甘油的能力,并通过易错PCR对导入的WS酶序列进行了随机突变,通过尼罗红荧光染色进行定向筛选,最终得到了三酰甘油积累量是突变前2.5倍的突变菌株。该方法展现了高通量筛选以及随机突变在酶工程中的应用潜力,可用于快速筛选其他可合成中性酯的酶,或者筛选对特定底物有活性的突变体。
3.3 应用于脂肪酸乙酯生产生物柴油可由长链脂肪酸和短链醇(主要是甲醇和乙醇)进行酯化反应产生,分子组成为脂肪酸甲酯和脂肪酸乙酯。相对于传统石油燃料,生物柴油具有可再生、无毒性、闪点高、易燃性低、使用安全性高等优点。如上文所述,WS具有极广的底物范围,这一性质被用来在各种重组宿主体内合成不同的生物柴油分子。对于脂肪酸乙酯的生产而言,最早被发现的WS的天然宿主A. baylyi是严格的需氧生物,其无法产生脂肪酸乙酯的重要前体乙醇,不是理想的生产菌株,因此,人们开始寻找其他工业菌株作为生产宿主。
2006年,Kalscheuer等[40]将丙酮酸脱羧酶基因(pdc)和乙醇脱氢酶基因(adhB)导入E. coli以构建乙醇合成途径,然后将来自A. baylyi ADP1的WS基因导入E. coli,重组菌株能够产生大量的乙醇并成功表达WS,但是E. coli自身产生的脂肪酸不足以支撑脂肪酸乙酯的合成,只有在外源添加大量脂肪酸的前提下才能正常生产脂肪酸乙酯。2015年,Röttig等[41]改造了E. coli脂肪酸合成途径以增加脂肪酸乙酯生产的前体,之后分别将不同来源的10种酰基转移酶基因在E. coli体内异源表达,发现将来自A. baylyi的WS进行点突变后得到的突变体AtfA(G355I)最适合脂肪酸乙酯的合成,最终产量可达到500 mg/L。
相对于E. coli,S. cerevisiae自身有着高效率的乙醇生产途径,且正常生长过程中积累的脂质主要含C16和C18链长的脂肪酸[42],这些脂肪酸很适合生产脂肪酸乙酯[43]。Yu等[10]于2012年将S. cerevisiae的甘油产乙醇途径和WS/DGAT产脂肪酸乙酯途径结合,在消耗17 g/L甘油的条件下得到0.52 g/L脂肪酸乙酯。2014年,Thompson等[44]将WS基因导入S. cerevisiae,然后利用多种方法,如过表达内源脂肪酸合成酶(FAS)以增强脂肪酸的合成途径,敲除β氧化等竞争途径基因以及对酰基转移酶基因进行密码子优化等,最终将S. cerevisiae自身的脂肪酸乙酯产量提高到25 mg/L。2015年,Eriksen等[45]将来自M. hydrocarbonoclasticus DSM 8798的WS基因和放线菌的Ⅰ型FAS酶基因导入S. cerevisiae,尝试在S. cerevisiae中引入异源的脂肪酸合成途径以进一步增加脂肪酸乙酯的产量,最终使产量相比未导入异源FAS酶基因时提高了6.3倍。
S. cerevisiae生产脂肪酸乙酯受到的限制主要在于它自身生产脂肪酸的能力较低,而一些产油酵母可能具有更好的生产潜力。它们能积累大量的脂质,最多可以达到其生物量的60%(质量分数),经过改造的工程化解脂耶氏酵母(Yarrowia lipolytica)产生的脂质产量高达72.7 g/L[46]。因此,Y. lipolytica等产油酵母可能具有非常大的脂肪酸乙酯生产潜力。Xu等[47]仅将过氧化物酶体/线粒体肉碱酰基转移酶perCat2过表达和WS异源表达相结合,得到的脂肪酸乙酯产量就达到142.5 mg/L。2018年,Gao等[48]将不同物种的WS基因经过密码子优化后在Y. lipolytica中异源表达,证明了来自M. hydrocarbonoclasticus DSM 8798菌株的WS基因生产脂肪酸乙酯的效率最高,之后他们通过敲除β-氧化、优化WS基因启动子等操作,最终使脂肪酸乙酯的产量达到了1.18 g/L,该酵母是目前报道的脂肪酸乙酯产量最高的重组酵母。
如上所述,在不同菌株中表达WS生产脂肪酸乙酯的研究中,产油酵母Y. lipolytica具有强大的脂肪酸积累能力和较强的脂肪酸乙酯生产能力,可能是生产脂肪酸乙酯等脂肪酸类生物产品的潜力巨大的菌株。
3.4 应用于脂肪酸短支链酯生产目前市场上的生物柴油多为脂肪酸甲酯和脂肪酸乙酯,研究表明相对于相同链长的直链酯,具有支链醇的脂肪酸酯(例如脂肪酸异丙酯、脂肪酸异丁酯)的倾点和浊点会降低,表现出良好的冷流性;同时支链酯的十六烷值也高于相同链长的直链酯,因此具有更好的燃料性能[49]。
相对于生产脂肪酸乙酯,微生物利用WS生产脂肪酸短支链酯(fatty acid short- and branched-chain esters, FASBEs)的研究较少。2006年Kalscheuer等[7]将来自A. baylyi ADP1的WS基因导入E. coli中,探究异源蜡酯的生物合成途径,意外地检测到脂肪酸丁酯的形成,他们将这一结果归因于培养基的胰蛋白胨中含有痕量的1-丁醇。这一结果证明WS的底物特异性很低,能够利用各种长度的醇类。2015年Tao等[50]尝试在E. coli中导入WS基因,同时结合E. coli支链脂肪酸合成途径与支链氨基酸生物合成途径,以提供异丁醇和3-甲基-1-丁醇前体,得到273 mg/L的脂肪酸支链酯。他们将同样的策略用于毕赤酵母(Pichia pastoris),得到169 mg/L的脂肪酸支链酯。在另一项研究中,Teo等[51]将2种蜡酯合成酶基因(来自Marinobacter sp.的WS2和Maqu_0168)导入S. cerevisiae,同时过表达S. cerevisiae内源的异丁醇途径酶,经优化得到产量达230 mg/L的脂肪酸短链酯,包括脂肪酸乙酯、脂肪酸丙酯、脂肪酸丁酯、脂肪酸异丁酯、脂肪酸异戊酯等。
上述研究利用合成生物学改造宿主使其提供短链支链醇,合成相应的脂肪酸短支链酯,展现了WS在生物能源领域中的巨大潜力,其不仅可以合成传统的生物燃料脂肪酸乙酯或脂肪酸甲酯,还可以合成性能更优的新型燃料分子。WS在酯类化合物生产中的应用的总结见表 3。
| 下载CSV 表 3 WS在酯类化合物生产中的应用 Table 3 Applications of wax ester synthases in production of ester compounds |
应用WS生产各种酯类的核心问题是如何改变酯类的化学成分以及如何提高酯类的产量,解决这些问题的关键在于提高WS的活性及改变它的特异性,目前对WS的优化主要也是朝着这个方向进行的。为了让工程菌株生产特定成分的酯类,可以对自然界中发现的大量WS进行筛选和表征,也可以通过蛋白质工程改造WS的最适底物,或者通过建立有效的筛选系统进行酶的定向进化以得到更适合生产目标酯类的酶[9, 39]。例如WS具有高度保守的HHxxxDG基序,该基序的随意替代会使酶的活性明显降低[27],将来自M. aquaeolei VT8的WS的部分位点进行突变后能改变其对醇类的选择性[28-29],对A. baylyi的蜡酯合成酶AtfA进行点突变得到的AtfA(G355I)则能够使其失去DGAT酶活性,同时提高脂肪酸乙酯的产量[41]。如何进一步改造WS使其更有利于生产应用将是未来研究的重点之一。
另一方面,WS在工程菌株中异源表达后,可通过代谢工程与合成生物学手段对宿主进行改造,通过改变前体调整合成的酯类化合物成分。例如,通过在工程菌中异源表达脂肪酰基还原酶,提供不同的脂肪醇前体,可以合成各种各样的蜡酯;通过组合不同来源的WS和不同来源的FAR,能够生产出种类更多的酯类化合物[52]。调节生物体内自身的脂肪酸组成也有助于生产不同种类的蜡酯,已经发现抑制本氏烟草(Nicotiana benthamiana)的KASII基因表达可以改变C16和C18酰基底物的比例,从而改变产生的蜡酯种类[53],这一思路可能对微生物生产不同种类蜡酯具有启发作用。
目前,利用工程菌株生产蜡酯的产量仍然没有达到工业化水平,其主要原因之一是脂肪酸或脂肪醇前体供应不足,常用的工业化菌株E. coli和S. cerevisiae自身的前体积累能力难以满足大量蜡酯的生产[45]。通过引入异源脂肪酸生产途径、敲除脂质的竞争途径等手段可以在一定程度上提高蜡酯的产量[37-38, 41, 45, 50], 这些方法在产量优化的思路上占据主导地位。另外,寻找其他适合蜡酯生产的工程菌株,例如脂质积累能力比E. coli和S. cerevisiae更强的产油酵母[46]具有良好的应用前景,目前报道的脂肪酸乙酯产量最高的工程菌株为非模式产油酵母Y. lipolytica[48]。
| [1] |
MARKUS R, LUKAS S. Protecting against water loss:analysis of the barrier properties of plant cuticles[J]. Journal of Experimental Botany, 2001, 52(363): 2023-2032. DOI:10.1093/jexbot/52.363.2023 |
| [2] |
KRAUSS P, MARKSTÄDTER C, RIEDERER M. Attenuation of UV radiation by plant cuticles from woody species[J]. Plant, Cell & Environment, 1997, 20(8): 1079-1085. |
| [3] |
KOLATTUKUDY P E. Chemistry and biochemistry of natural waxes[M]. Amsterdam: Elsevier, 1976.
|
| [4] |
CHENG J B, RUSSELL D W. Mammalian wax biosynthesis:Ⅱ. expression cloning of wax synthase cDNAs encoding a member of the acyltransferase enzyme family[J]. The Journal of Biological Chemistry, 2004, 279(36): 37798-37807. DOI:10.1074/jbc.M406226200 |
| [5] |
STEINBVCHEL A. Perspectives for biotechnological production and utilization of biopolymers:metabolic engineering of polyhydroxyalkanoate biosynthesis pathways as a successful example[J]. Macromolecular Bioscience, 2001, 1(1): 1-24. DOI:10.1002/1616-5195(200101)1:1<1::AID-MABI1>3.0.CO;2-B |
| [6] |
IVEN T, HORNUNG E, HEILMANN M, et al. Synthesis of oleyl oleate wax esters in Arabidopsis thaliana and Camelina sativa seed oil[J]. Plant Biotechnology Journal, 2016, 14(1): 252-259. DOI:10.1111/pbi.12379 |
| [7] |
KALSCHEUER R, STÖVEKEN T, LUFTMANN H, et al. Neutral lipid biosynthesis in engineered Escherichia coli:jojoba oil-like wax esters and fatty acid butyl esters[J]. Applied and Environmental Microbiology, 2006, 72(2): 1373-1379. DOI:10.1128/AEM.72.2.1373-1379.2006 |
| [8] |
ISHIGE T, TANI A, SAKAI Y, et al. Wax ester production by bacteria[J]. Current Opinion in Microbiology, 2003, 6(3): 244-250. DOI:10.1016/S1369-5274(03)00053-5 |
| [9] |
RÖTTIG A, STEINBVCHEL A. Random mutagenesis of AtfA and screening for Acinetobacter baylyi mutants with an altered lipid accumulation[J]. European Journal of Lipid Science and Technology, 2013, 115(4): 394-404. DOI:10.1002/ejlt.201200401 |
| [10] |
YU K O, JUNG J, KIM S W, et al. Synthesis of FAEEs from glycerol in engineered Saccharomyces cerevisiae using endogenously produced ethanol by heterologous expression of an unspecific bacterial acyltransferase[J]. Biotechnology and Bioengineering, 2012, 109(1): 110-115. DOI:10.1002/bit.23311 |
| [11] |
LI F L, WU X M, LAM P, et al. Identification of the wax ester synthase/acyl-coenzyme A:diacylglycerol acyltransferase WSD1 required for stem wax ester biosynthesis in Arabidopsis[J]. Plant Physiology, 2008, 148(1): 97-107. DOI:10.1104/pp.108.123471 |
| [12] |
KALSCHEUER R, STEINBVCHEL A. A novel bifunctional wax ester synthase/acyl-CoA:diacylglycerol acyltransferase mediates wax ester and triacylglycerol biosynthesis in Acinetobacter calcoaceticus ADP1[J]. The Journal of Biological Chemistry, 2003, 278(10): 8075-8082. DOI:10.1074/jbc.M210533200 |
| [13] |
REISER S, SOMERVILLE C. Isolation of mutants of Acinetobacter calcoaceticus deficient in wax ester synthesis and complementation of one mutation with a gene encoding a fatty acyl coenzyme A reductase[J]. Journal of Bacteriology, 1997, 179(9): 2969-2975. DOI:10.1128/JB.179.9.2969-2975.1997 |
| [14] |
STÖVEKEN T, KALSCHEUER R, MALKUS U, et al. The wax ester synthase/acyl coenzyme A:diacylglycerol acyltransferase from Acinetobacter sp. strain ADP1:characterization of a novel type of acyltransferase[J]. Journal of Bacteriology, 2005, 187(4): 1369-1376. DOI:10.1128/JB.187.4.1369-1376.2005 |
| [15] |
HOLTZAPPLE E, SCHMIDT-DANNERT C. Biosynthesis of isoprenoid wax ester in Marinobacter hydrocarbonoclasticus DSM 8798:identification and characterization of isoprenoid coenzyme A synthetase and wax ester synthases[J]. Journal of Bacteriology, 2007, 189(10): 3804-3812. DOI:10.1128/JB.01932-06 |
| [16] |
TOMIYAMA T, KURIHARA K, OGAWA T, et al. Wax ester synthase/diacylglycerol acyltransferase isoenzymes play a pivotal role in wax ester biosynthesis in Euglena gracilis[J]. Scientific Reports, 2017, 7: 13504. DOI:10.1038/s41598-017-14077-6 |
| [17] |
CHENG J B, RUSSELL D W. Mammalian wax biosynthesis:Ⅰ. identification of two fatty acyl-coenzyme A reductases with different substrate specificities and tissue distributions[J]. The Journal of Biological Chemistry, 2004, 279(36): 37789-37797. DOI:10.1074/jbc.M406225200 |
| [18] |
STURTEVANT D, LU S P, ZHOU Z W, et al. The genome of jojoba (Simmondsia chinensis):a taxonomically isolated species that directs wax ester accumulation in its seeds[J]. Science Advances, 2020, 6(11): 3240. DOI:10.1126/sciadv.aay3240 |
| [19] |
LARDIZABAL K D, METZ J G, SAKAMOTO T, et al. Purification of a jojoba embryo wax synthase, cloning of its cDNA, and production of high levels of wax in seeds of transgenic Arabidopsis[J]. Plant Physiology, 2000, 122(3): 645-655. DOI:10.1104/pp.122.3.645 |
| [20] |
SHI S B, VALLE-RODRIGUEZ J O, KHOOMRUNG S, et al. Functional expression and characterization of five wax ester synthases in Saccharomyces cerevisiae and their utility for biodiesel production[J]. Biotechnology for Biofuels, 2012, 5: 7. DOI:10.1186/1754-6834-5-7 |
| [21] |
RÖTTIG A, WOLF S, STEINBVCHEL A. In vitro characterization of five bacterial WS/DGAT acyltransferases regarding the synthesis of biotechnologically relevant short-chain-length esters[J]. European Journal of Lipid Science and Technology, 2015, 118(1): 124-132. |
| [22] |
MIKLASZEWSKA M, DITTRICH-DOMERGUE F, BANAS A, et al. Wax synthase MhWS2 from Marinobacter hydrocarbonoclasticus:substrate specificity and biotechnological potential for wax ester production[J]. Applied Microbiology and Biotechnology, 2018, 102(9): 4063-4074. DOI:10.1007/s00253-018-8878-8 |
| [23] |
ZHANG N N, MAO Z J, LUO L, et al. Two bifunctional enzymes from the marine protist Thraustochytrium roseum:biochemical characterization of wax ester synthase/acyl-CoA:diacylglycerol acyltransferase activity catalyzing wax ester and triacylglycerol synthesis[J]. Biotechnology for Biofuels, 2017, 10: 185. DOI:10.1186/s13068-017-0869-y |
| [24] |
MIKLASZEWSKA M, ADAM K, ANTONI B. Detailed characterization of the substrate specificity of mouse wax synthase[J]. Acta Biochimica Polonica, 2013, 60(2): 209-215. |
| [25] |
BIESTER E M, HELLENBRAND J, GRUBER J, et al. Identification of avian wax synthases[J]. BMC Biochemistry, 2012, 13(1): 4. DOI:10.1186/1471-2091-13-4 |
| [26] |
KING A, NAM J W, HAN J, et al. Cuticular wax biosynthesis in petunia petals:cloning and characterization of an alcohol-acyltransferase that synthesizes wax-esters[J]. Planta, 2007, 226(2): 381-394. DOI:10.1007/s00425-007-0489-z |
| [27] |
STÖVEKEN T, KALSCHEUER R, STEINBVCHEL A. Both histidine residues of the conserved HHXXXDG motif are essential for wax ester synthase/acyl-CoA:diacylglycerol acyltransferase catalysis[J]. European Journal of Lipid Science and Technology, 2009, 111(2): 112-119. DOI:10.1002/ejlt.200800167 |
| [28] |
BARNEY B M, MANN R L, OHLERT J M. Identification of a residue affecting fatty alcohol selectivity in wax ester synthase[J]. Applied and Environmental Microbiology, 2013, 79(1): 396-399. DOI:10.1128/AEM.02523-12 |
| [29] |
BARNEY B M, OHLERT J M, TIMLER J G, et al. Altering small and medium alcohol selectivity in the wax ester synthase[J]. Applied Microbiology and Biotechnology, 2015, 99(22): 9675-9684. DOI:10.1007/s00253-015-6783-y |
| [30] |
PETRONIKOLOU N, NAIR S. Structural and biochemical studies of a biocatalyst for the enzymatic production of wax esters[J]. ACS Catalysis, 2018, 8(7): 6334-6344. DOI:10.1021/acscatal.8b00787 |
| [31] |
LEHTINEN T, EFIMOVA E, SANTALA S, et al. Improved fatty aldehyde and wax ester production by overexpression of fatty acyl-CoA reductases[J]. Microbial Cell Factories, 2018, 17(1): 19. DOI:10.1186/s12934-018-0869-z |
| [32] |
FIXTER L M, NAGI M N, MCCORMACK J G, et al. Structure, distribution and function of wax esters in Acinetobacter calcoaceticus[J]. Microbiology, 1986, 132(11): 3147-3157. DOI:10.1099/00221287-132-11-3147 |
| [33] |
PAZYAR N, YAGHOOBI R, GHASSEMI M R, et al. Jojoba in dermatology:a succinct review[J]. Giornale Italiano di Dermatologia e Venereologia, 2013, 148(6): 687-691. |
| [34] |
VRKOSLAV V, URBANOVÁ K, HÁKOVÁ M, et al. Analysis of wax esters by silver-ion high-performance liquid chromatography-tandem mass spectrometry[J]. Journal of Chromatography A, 2013, 1302: 105-110. DOI:10.1016/j.chroma.2013.06.031 |
| [35] |
OH C S, TOKE D A, MANDALA S, et al. ELO2 and ELO3, homologues of the Saccharomyces cerevisiae ELO1 gene, function in fatty acid elongation and are required for sphingolipid formation[J]. The Journal of Biological Chemistry, 1997, 272(28): 17376-17384. DOI:10.1074/jbc.272.28.17376 |
| [36] |
TEERAWANICHPAN P, QIU X. Fatty acyl-CoA reductase and wax synthase from Euglena gracilis in the biosynthesis of medium-chain wax esters[J]. Lipids, 2010, 45(3): 263-273. DOI:10.1007/s11745-010-3395-2 |
| [37] |
WENNING L, YU T, DAVID F, et al. Establishing very long chain fatty alcohol and wax ester biosynthesis in Saccharomyces cerevisiae[J]. Biotechnology and Bioengineering, 2017, 114(5): 1025-1035. DOI:10.1002/bit.26220 |
| [38] |
WENNING L, EJSING C S, DAVID F, et al. Increasing jojoba-like wax ester production in Saccharomyces cerevisiae by enhancing very long-chain, monounsaturated fatty acid synthesis[J]. Microbial Cell Factories, 2019, 18(1): 49. DOI:10.1186/s12934-019-1098-9 |
| [39] |
SANTÍN O, GALIÉ S, MONCALIÁN G. Directed evolution of a bacterial WS/DGAT acyltransferase:improving tDGAT from Thermomonospora curvata[J]. Protein Engineering, Design and Selection, 2019, 32(1): 25-32. |
| [40] |
KALSCHEUER R, STOLTING T, STEINBUCHEL A. Microdiesel:Escherichia coli engineered for fuel production[J]. Microbiology, 2006, 152(9): 2529-2536. DOI:10.1099/mic.0.29028-0 |
| [41] |
RÖTTIG A, ZUREK P J, STEINBVCHEL A. Assessment of bacterial acyltransferases for an efficient lipid production in metabolically engineered strains of E. coli[J]. Metabolic Engineering, 2015, 32: 195-206. DOI:10.1016/j.ymben.2015.09.016 |
| [42] |
TROTTER P J. The genetics of fatty acid metabolism in Saccharomyces cerevisiae[J]. Annual Review of Nutrition, 2001, 21: 97-119. DOI:10.1146/annurev.nutr.21.1.97 |
| [43] |
WESTFALL P J, GARDNER T S. Industrial fermentation of renewable diesel fuels[J]. Current Opinion in Biotechnology, 2011, 22(3): 344-350. DOI:10.1016/j.copbio.2011.04.023 |
| [44] |
THOMPSON R A, TRINH C T. Enhancing fatty acid ethyl ester production in Saccharomyces cerevisiae through metabolic engineering and medium optimization[J]. Biotechnology and Bioengineering, 2014, 111(11): 2200-2208. DOI:10.1002/bit.25292 |
| [45] |
ERIKSEN D T, HAMEDIRAD M, YUAN Y B, et al. Orthogonal fatty acid biosynthetic pathway improves fatty acid ethyl ester production in Saccharomyces cerevisiae[J]. ACS Synthetic Biology, 2015, 4(7): 808-814. DOI:10.1021/sb500319p |
| [46] |
XU P, QIAO K J, STEPHANOPOULOS G. Engineering oxidative stress defense pathways to build a robust lipid production platform in Yarrowia lipolytica[J]. Biotechnology and Bioengineering, 2017, 114(7): 1521-1530. DOI:10.1002/bit.26285 |
| [47] |
XU P, QIAO K J, AHN W S, et al. Engineering Yarrowia lipolytica as a platform for synthesis of drop-in transportation fuels and oleochemicals[J]. Proceedings of the National Academy of Sciences, 2016, 113(39): 10848-10853. DOI:10.1073/pnas.1607295113 |
| [48] |
GAO Q, CAO X, HUANG Y Y, et al. Overproduction of fatty acid ethyl esters by the oleaginous yeast Yarrowia lipolytica through metabolic engineering and process optimization[J]. ACS Synthetic Biology, 2018, 7(5): 1371-1380. DOI:10.1021/acssynbio.7b00453 |
| [49] |
RÖTTIG A, WENNING L, BROKER D, et al. Fatty acid alkyl esters:perspectives for production of alternative biofuels[J]. Applied Microbiology and Biotechnology, 2010, 85(6): 1713-1733. DOI:10.1007/s00253-009-2383-z |
| [50] |
TAO H, GUO D Y, ZHANG Y C, et al. Metabolic engineering of microbes for branched-chain biodiesel production with low-temperature property[J]. Biotechnology for Biofuels, 2015, 8: 92. DOI:10.1186/s13068-015-0270-7 |
| [51] |
TEO W S, LING H, YU A Q, et al. Metabolic engineering of Saccharomyces cerevisiae for production of fatty acid short- and branched-chain alkyl esters biodiesel[J]. Biotechnology for Biofuels, 2015, 8: 177. DOI:10.1186/s13068-015-0361-5 |
| [52] |
YU D, HORNUNG E, IVEN T, et al. High-level accumulation of oleyl oleate in plant seed oil by abundant supply of oleic acid substrates to efficient wax ester synthesis enzymes[J]. Biotechnology for Biofuels, 2018, 11: 53. DOI:10.1186/s13068-018-1057-4 |
| [53] |
ASLAN S, HOFVANDER P, DUTTA P, et al. Transient silencing of the KASII genes is feasible in Nicotiana benthamiana for metabolic engineering of wax ester composition[J]. Scientific Reports, 2015, 5: 11213. DOI:10.1038/srep11213 |








